Polimorfismos del sistema HLA (loci A*, B* y DRB1*) en población colombiana
Humberto Ossa1, Andrea Manrique1, Sonia Quintanilla1, Alejandro Peña2
1Laboratorio de Genética y Biología Molecular.
2Universidad Distrital Francisco José de Caldas. Proyecto Curricular Licenciatura en Biología
Correspondencia: hossa@cable.net.co
Recibido: 20-03-2007 / Aceptado: 02-04-2007
Resumen
El objetivo de este trabajo fue determinar las frecuencias alélicas del sistema HLA-A*,B* y DRB1* en una muestra de 981 individuos de la población colombiana. El tamaño de la muestra fue depurado a partir de la base de datos del la Red Colombiana de Trasplantes de donde se tomaron finalmente 981 individuos a los cuales se les realizó el análisis estadístico por medio del programa ARLEQUIN versión 2000. Se encontró una diversidad total de 66 alelos que representan el 87% de la diversidad alélica mundial a nivel de los loci A*, B* y DRB1*. Se hallaron 19 alelos para el locus HLA-A*, 33 para el locus HLA-B* y 14 para el DRB1*. Los alelos HLA-A*02 y HLA-A*24 presentaron una frecuencia de 0,2696 y 0,2706 respectivamente, que corresponden al 54% de la muestra analizada.
Los alelos HLA-B*35 y HLA-B*51 fueron los más frecuentes del loci B*: 0,1926 y 0,1314, respectivamente. Para los DRB1*, únicamente el DRB1*04 presenta una alta frecuencia: 0.238. Los restantes alelos muestran frecuencias inferiores al 12%. Los resultados de este trabajo confirman, a nivel molecular, la diversidad genética de la población estudiada, dado que Colombia posee el 87% de los polimorfismos HLA (loci A*, B* y DRB1*) encontrados en el mundo.
Palabras Clave: alelos, antígeno mayor de histocompatibilidad, frecuencia alélica, haplotipo, polimorfismo genético, trasplante.
Abstract
The objective of this work was to determine the allelic frequencies of system HLA-A*, B* and DRB1* in a sample of 981 individuals of the Colombian population. The sample size was purified from the data base of the Colombian Network of transplantation from which 981 individuals were taken to apply the statistical analysis to them by means of program ARLEQUIN version 2000. A total diversity of 66 alleles was found. They represent 87% of world-wide allelic diversity level of loci A*, B* and DRB1*. 19 alleles for locus HLA-A*, 33 for locus HLA-B* and 14 for the DRB1* were found. Alleles HLA A*02 and HLA-A*24 respectively displayed a frequency of 0.2696 and 0.2706, that correspond to 54% of the analyzed sample.
Alleles HLA-B*35 and HLA-B*51 were the most frequent of loci B* were 0,1926 and 0.1314, respectively. For the DRB1*, the DRB1*04 presents a high frequency solely: 0.238. The left over alleles show inferior frequencies 12%. The results of this work confirm, at a molecular level, the genetic diversity of the Colombian population given that Colombia has 87% of polymorphisms HLA (loci A*, B* and DRB1*) found in the world.
Key words: alleles, allelic frequency, Major Histocompatibility Complex, haplotype, polymorphism genetic, transplantation.
Introducción
Los genes que codifican para el sistema de antígenos de leucocitos humanos (HLA), por sus siglas en inglés, son muy polimórficos y además, los más ampliamente estudiados en la especie humana (1). Estos genes están ligados y forman el complejo mayor de histocompatibilidad (CMH) que ocupa aproximadamente 4000 kb del brazo corto del cromosoma 6 (6p21.1 - 6p21.3 (2).
Son los encargados de dirigir funciones muy importantes dentro de la homeostasis del organismo, que van desde la regulación de la respuesta inmune celular y humoral, pasando por la presentación de antígenos de patógenos intra-celulares a los linfocitos T citotóxicos (LTc) encargados de la respuesta inmune efectora; hasta ser la fuente de adaptación más importante con los microorganismos que nos rodean e interactuamos a diario y cuya gran diversidad y cambio constante ha inducido a este sistema genético.
Toda esta gama de funciones fisiológicas que cumplen las moléculas HLA hacen que se vean asociadas con enfermedades, especialmente con las de tipo autoinmune, de tal manera que la respuesta del sistema inmune a la infección es, al menos en parte, dependiente de las diferentes combinaciones de la herencia del sistema (3).
En lo referente a la aceptación o rechazo de tejido alogénico trasplantado, la función del sistema HLA está ampliamente documentada y aceptada por la comunidad científica (4). Es un hecho que a mayor compatibilidad, mayor será la aceptación del tejido trasplantado, llegando a incrementar hasta en un 20% el éxito en trasplante renal a más de 5 años, aspecto muy importante a tener en cuenta por los centros que realizan trasplantes en todo el país (5). El presente estudio está diseñado para determinar las frecuencias alélicas de los loci HLAA*, B* y DRB1* en una muestra de 981 colombianos.
Materiales y métodos
Para el estudio de las frecuencias alélicas se tomó una muestra final de 981 individuos no relacionados, tipificados para los loci HLA A*, B* y DRB1*. La muestra estuvo compuesta tanto de receptores como de donantes para trasplante renal y médula ósea, todos pertenecientes a la base de datos de la red colombiana de trasplantes integrada por la Clínica de Marly, Hospital Universitario San Ignacio, Hospital San José, Clínica San Rafael y la Fundación Cardioinfantil. Estas muestras, provenientes de todo el país, fueron enviadas para tipificación HLA pre-trasplante.
Este trabajo se inició en Enero de 2004, comenzando con la depuración de la base de datos de acuerdo a los siguientes criterios de inclusión: a) Historia clínica completa, que incluye copia de la planilla de tipificación, mostrando un resultado verificado manualmente y mediante el software suministrado por las casas comerciales; b) Fotografía del gel en medio físico o digital; c) Copia del informe final reportado a la red colombiana de trasplantes y d) Amplificación clara de los antígenos públicos.
Las frecuencias alélicas de los loci HLA A*, B* y DRB1* se obtuvieron con el Programa Arlequín 2000 (7). Los algoritmos utilizados por este software son los siguientes: a) frecuencias alélicas q = çi/N, b) varianza: V(q) = [q-(1-q)]/N, c) desviación estándar: sd=v[V(q)]². El equilibrio de Hardy Weinberg en cada uno de los locus (8, 9) se calculó con la siguiente formula. ?Pi=1, Pi= [Pii +?(Pii/2)] = (2Nij/2N) + ?(Nii/2N)
Resultados
El análisis de los datos para Colombia arrojó un total de 66 polimorfismos HLA, distribuidos así: 19 alelos para el locus A*, 33 para el locus B* y 14 para el locus DRB1*. A nivel mundial se han reportado 22 polimorfismos para el HLA-A*, 40 para el HLAB* y 14 para HLADRB1*. Esta nomenclatura corresponde a especificidades serológicas y presenta los polimorfismos HLA bajo la resolución de dos dígitos.
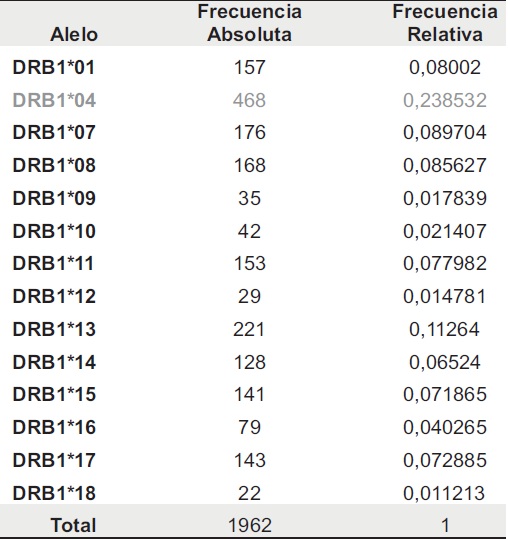
Los resultados obtenidos muestran la diversidad genética de la población colombiana. El locus HLAA*, con 19 alelos, es el único locus que no presenta equilibrio de Hardy-Weinberg (EHW). Esta pérdida del equilibrio se debe a la distribución no homogénea de las frecuencias de los diferentes alelos, donde el HLA*02 y HLA*24 presentan alta frecuencia (0,269623 y 0,270642), respectivamente, es decir, el 54% de los colombianos estudiados portan estos alelos. El resto de los alelos HLA-A*, 17 en total, se encuentran en una frecuencia menor del 7% (Tabla 1). El HLA-A*02, al igual que todas las poblaciones mundiales estudiadas, presenta alta frecuencia en la población colombiana (10-13).
Tabla 1. Frecuencias alélicas del locus HLA-A*. 2n = 1962
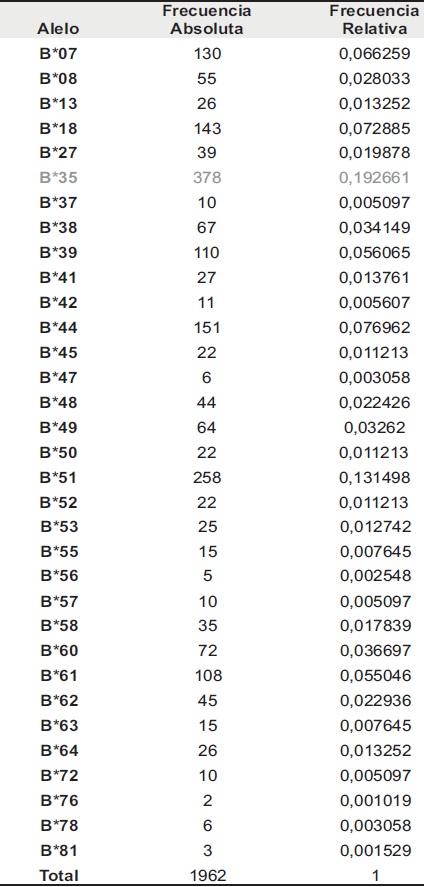
De los 33 alelos encontrados para el locus HLA-B*, el HLA-B*35 y el HLA-B*51, fueron los más frecuentes: 0,192661 y 0,131498 respectivamente. Los demás alelos, 31 en total, presentaron frecuencias inferiores al 8%, Tabla 2.
Tabla 2. Frecuencias alélicas del locus HLA-B*. 2n= 1962
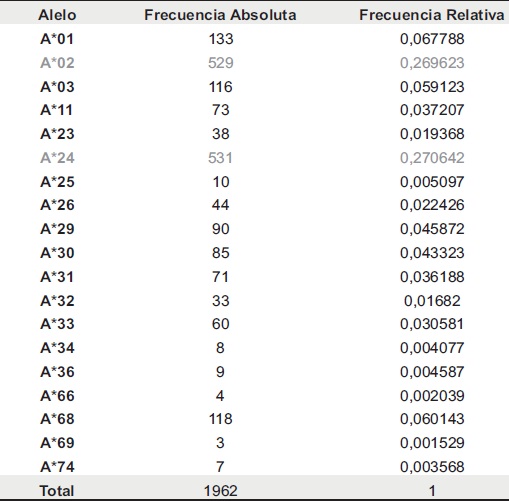
El polimorfismo del locus HLA-DRB1* está representado por 14 alelos, que constituyen la totalidad de polimorfismos para este locus a nivel mundial. El HLADRB1*04 y el DRB1*13 exhibieron las frecuencias más altas: 0.2385 y 0.1126 respectivamente, seguidos por DRB1*08 y DRB1*07 con 0.0856 y 0.0897 respectivamente (Tabla 3). Los demás alelos presentaron frecuencias aún más bajas. Este locus se encuentra en EHW.
Tabla 3. Frecuencias alélicas del locus HLA-DRB1*. 2n= 1962
Discusión
Los resultados obtenidos muestran el alto grado de polimorfismo del sistema HLA en los tres locus estudiados. Se encontraron 66 de los 76 grupos alélicos conocidos para el HLA A*, B* y DRB1* a nivel mundial. Para el caso del locus HLA-A* se observó una distribución desproporcionada de las diferentes frecuencias alélicas, donde A*02 y A*24 representan el 54% de la diversidad alélica de este locus. Bera y colaboradores (14) encontraron en una muestra de Francia de 200 individuos, una frecuencia similar para A*02 de 0.27 y en A*24 de 0.24; Pablo de R. y col. (15) reportan una frecuencia para HLA* 02 de 0.54 y en A*24 de 0.1038 en 148 individuos peruanos; Cao K y colaboradores (16) muestran que A*02 es muy frecuente en poblaciones caucásicas, nativos norte americanos e hispanos. Estos datos son similares a los resultados encontrados en la población colombiana analizada en este trabajo.
El comportamiento del locus B* es similar al encontrado en el locus A*, pero tiene una distribución de frecuencias más homogénea. El locus DRB1* es el que presenta mayor homogeneidad en las frecuencias de todo el sistema. En lo referente a trasplante de órganos, los resultados de este trabajo deben convertirse en la base para clasificar los pacientes en lista de espera de la red nacional de trasplantes, de tal manera que se agrupen aquellos pacientes que portan alelos frecuentes y que se espera consigan donantes efectivos rápidamente; mientras que, aquellos que porten alelos poco frecuentes tendrán menos probabilidad de resolver su situación, llegando, casi siempre, a tener que esperar varios años en diálisis, engrosando lista de espera y elevando los costos de sostenimiento.
Además, estos pacientes tienen derecho a saber el motivo por el cual no logran conseguir un donante efectivo. Los datos presentados en este trabajo permiten agrupar todos los receptores inscritos en la red nacional de trasplantes de acuerdo a la frecuencia de sus alelos HLA. Estos datos representan un panorama general de la distribución de frecuencias estimadas del sistema HLA en Colombia.
Agradecimientos.
Dr. Alexis Bolívar - Química Internacional, Dr. Guillermo Aldana, Dr. Humberto Bohórquez, Dr. Carlos Andrés Benavides, por remitir los pacientes que hacen parte de la muestra. A la Dra. María Paula Gómez, directora de la red nacional de trasplantes, por su apoyo y confianza y al Dr. Oscar Quiroz, Hospital San Vicente de Paul – Medellín, por su aporte en el desarrollo del proyecto durante su entrenamiento en Bogotá.
Referencias
1. MHC Sequencing Consortium. Complete sequence and gene map of a human major histocompatibility complex. MHC Sequencing Consortium. Nature. 1999;401:921-923.
2. Hardy H. Mendelian Proportions in a mixed population. Science. 1908;28:49-50.
3. Benjamin A, Bradley T. Pronostic assays for rejection and tolerance in organ transplantation. Transplant Immunol. 2005;14:193-201.
4. Ober C, Weitkamp L, Cox N, Dytch H, Kostyu D, Elias S. HLA and mate choice in humans. Am J. Hum. Genet. 1997;61:497-504.
5. Halpern S, Ubel P, Caplan A. Solid-organ trasplantation in HIV-infected patients. N Engl J Med. 2002;347:284-287.
6. www.ncbi.nlm.nih.gov/projects/dbmhc
7. Stefan S, Scheneider D, Excoffier L. Arlequin 2.000: a Software for population genetics data analysis. 2000. Genetics and Biometry Labs, University of Genera, Switzerland.
8. Matsuura M, Eguchi S. Estimation of gene frequency and test for Hardy-Weinberg Equilibrium in the HLA system. Environ Health Perspect. 1990;87:149-155.
9. Robinson J, Waller M, Parham P, Groot N, Bontrop R, Kennedy L, Stoehr P, Marsh S. E.IMGT/HLA and IMGT MHC: Sequence databases for the study of the major histocompatibility complex. Nucleic Acids Research. 2003;31:311-314.
10. Ossa H, Ramos O, Yunis E. Estudios genéticos de las comunidades indígenas del nororiente colombiano. Rev. Facultad de Medicina Universidad Nacional. 1994;42:9-16.
11. Olerup O, Zetterquist H. HLA-DR Typing by PCR amplification with sequence specific primer (PCR SSP) In 2 hours: an alternative to serological DR typing in clinical practice including donor-recipient matching in cadaveric transplantations. Tissue Antigens. 1992;39:225-235.
12. Crespí C, Milà J, Martínez-Pomar N, Etxagibel A, Muñoz- Saa I, Priego D, Luque A, Pons J, Picornell A, Ramon M, Castro J, Matamoros N. HLA polymorphism in a Majorcan Population of Jewish Descent: Comparision with Majorca, Minorca, Ibiza (Balearic Islands) and other jewish communities. Tissue Antigens. 2002;60:282-291.
13. Garrigan D, Hendrick P. Perspective: Detecting adaptative molecular polymorphism: Lessons from de MHC. Evolution Int J Org Evolution. 2003;57: 1701-1722.
14. Bera, O, Cesaire R, Quelvennec E, HLA class I and class II allele and haplotype diversity in Martinicans, Tissue Antigens. 2001;57:200-207
15. De Pablo R, Beraun Y, Nieto A., Calzada J, Rementeria M, Sanz L, López-Nevot M, Martín J. - HLA class I and class II allele distribution in the Peruvian population. Tissue Antigens, 2000;56:507-514.
16. Cao K, Hollenbach J, Shi X, Shi W, Chopek M, Fernández-Viña MAAnalysis of the frequencies of HLA A, B, and C alleles and haplotypes in the five major ethnic groups of the United States reveals high levels of diversity in these loci and contrasting distribution patterns in these populations. Hum Immunol. 200;62:1009 1030.



