Memorias del III Simposio Nacional y I Internacional de Actualización e Investigación en Ciencias Biomédicas
La Revista NOVA en formato electrónico publica los resúmenes de las conferencias magistrales de los módulos de genética, bioquímica, bioinformática, microbiología, parasitología, bioética y los resúmenes de los trabajos libres.
En formato impreso se encontrarán las memorias así:
El evento se llevó acabo en el mes de agosto de 2005 y fue organizado por la Universidad de la Sabana, la Universidad Colegio Mayor de Cundinamarca, la Universidad de Los Andes, la Universidad INCCA de Colombia, la Asociación Colombiana de Genética Humana – Grupo de Mutagénesis y Carcinogénesis y la Asociación Colombiana de Ciencias Biológicas.
Información: revistanova@unicolmayor.edu.co
Diana Torres López1, Giancarlo Ramelli Prati, Ignacio Briceño Balcázar1, Angela Umaña, Patricia Bond2, Ann Curtis, Saúl Rugeles1, Mauricio Tawil1, Lilian Torregrosa1, Ute Hamman3.
1Pontificia Universidad Javeriana.
2Institute of Human Genetics, University Of Newcastle Upon Tyne, Inglaterra.
3Ute Hamman, DKFC Alemania
Correspondencia: ignaciobb@unisabana.edu.co
Palabras claves: cáncer, genética, genes BRCA1 y BRCA2, mutación, Colombia.
Resumen
Mutaciones germinales en los genes BRCA1 y BRCA2 predisponen a los individuos a presentar cáncer de mseno, ovario y próstata. Realizamos un estudio mutacional en 54 casos índice pertenecientes a 54 familias que cumplieron los criterios de inclusión para cáncer familiar. Se utilizaron las técnicas SSCP/HA, PTT, HPLC y secuenciación directa. Detectamos mutaciones en trece familias. Tres de estas mutaciones se repitieron en varios casos; dos de BRCA1 se encontraron en tres familias y una en BRCA2 en una familia. Estudios de haplotipo demostraron la presencia de mutaciones fundadoras, reflejo de los procesos del poblamiento colombiano.
Estos resultados nos indican que en nuestro medio, al igual que en otros países, las mutaciones germinales en estos genes tienen una participación importante en las familias con síndrome de cáncer de seno y ovario.
Las técnicas utilizadas mostraron un balance costo beneficio favorable. Este trabajo muestra la pertinencia y viabilidad de llevar a cabo una investigación a gran escala en nuestro medio con la posibilidad de ofrecer un servicio diagnóstico.
Dr. Hugo A. Barrera Saldaña. Facultad de Medicina de la Universidad Autónoma de Nuevo León. Av. Madero y Dr. Eduardo Aguirre P. S/N.
Col. Mitras Centro, C.P. 64460, Monterrey, Nuevo León. México.
Correspondencia: hbarrera@fm.uanl.mx
Palabras clave: VPH, metilación de promotores, CaCu, alteraciones epigenéticas.
Resumen
El cáncer cérvico-uterino (CaCu) es causado por un cambio en la células de la pared del cervix (la parte baja del útero ó canal del nacimiento) que se transforman gradualmente a precancerosas. Las células pueden eventualmente convertirse en células cancerosas. Sin embargo, más del 50% de las mujeres con este tipo de lesiones logran regresar a la normalidad. El CaCu no presenta síntomas en sus estados tempranos, por lo que solo es detectado hasta sus estados más avanzados y por ende de mayor riesgo. En el año 2000, se estimó una incidencia de 470, 606 casos y 233,372 muertes debidas a CaCU en mujeres a nivel mundial, con más del 80% de estos valores siendo aportados por países en vías de desarrollo.
El CaCU es la principal causa de muerte por cáncer en mujeres de México, con cerca de 5000 muertes por año en la población femenina. El VPH ha sido encontrado en el 99.7% de los casos de CaCU a nivel mundial.
La magnitud de la asociación entre el HPV y el CaCU es más grande que la del tabaco con el cáncer de pulmón. Se estima que el 20% de la población femenina en México esta infectada con este virus, más de 100 tipos de VPHs han sido reconocidos en base a las diferencias que exhiben en su secuencia de ADN. Basados
en su asociación con el CaCU o las lesiones precursoras, los VPHs se han agrupado tanto en VPHs de alto riesgo, como de bajo riesgo. Los primeros incluyen al 16, 18, 31, 33, 34, 35, 39, 45, 51, 52, 56, 58, 59, 66, 68 y al 70, mientras que los últimos incluyen al 6, 11, 42 y 44. Aun cuando el porcentaje de esta infección es alta, no más del 2% de estas infecciones progresan a CaCU. Debido a ello, hay muchas preguntas acerca de la eliminación o persistencia de la infección y la progresión a CaCU.
Dentro de los factores asociados al riesgo de desarrollar cáncer por VPH, se han descrito alteraciones genéticas y/o epigenéticas. Las alteraciones genéticas son todas aquellas que cambian la estructura de un gen y que por lo tanto aumentan o disminuyen la actividad de la proteína codificada. Por su parte, las alteraciones epigenéticas no producen un cambio en la secuencia del gen, pero sí alteran el grado de su expresión y por ende los niveles de la proteína correspondiente. La principal alteración epigenética conocida y estudiada es la metilación del DNA en los promotores de genes, lo que inhibe su expresión. Estos factores se están investigando tanto en el genoma del virus como en el del huésped.
Alteraciones genéticas. Las diferencia entre un tipo de VPH y otro radica en las variaciones nucleotidicas en sus secuencias. Estos cambios afectan la actividad de las proteínas virales incluídas sus oncoproteínas. Es por esta razón que la atención reciente se ha concentrado en las marcadas diferencias entre el riesgo por virus encontrados en los carcinomas versus los encontrados en las infecciones transitorias.
Por lo que a genes celulares respecta, estudios experimentales han indicado que la proteína p53 con un residuo de arginina en la posición 72 es fácilmente degradada por la proteína E6 del VPH, sugiriendo que las personas homozigotas para el alelo Arg-Arg presentan una mayor susceptibilidad hacia el CaCU. De la misma forma, dado que la respuesta inmune del huésped hacia el VPH es una determinante en la persistencia del virus y la progresión a lesiones de alto grado y CaCU, es razonable pensar que las variaciones en HLA puedan afectar la patogénesis de la enfermedad a través del control inmunológico sobre el virus.
Alteraciones epigenéticas. La hipermetilación aberrante en las regiones rica en CpG del promotor de muchos genes supresores de tumores disminuye su transcripción y contribuye a la formación y progresión del CaCU. Igualmente, se especula que la hipometilación de la región promotora del genoma viral pueda potenciar la actividad viral y por ende su oncogenicidad.
Enfoque de nuestra investigación. En un tamizaje realizado en nuestra ciudad en el verano del 2002 para detectar la presencia del VPH, a partir de 4000 mujeres aparentemente sanas 600 resultaron positivas. A tres años de distancia, nos encontramos volviendo a contactar a estas últimas mujeres para indagar el estado de la infección, para ver si progresó o no hacia una condición precancerosa, o peor aún hacia una cancerosa. Nos proponemos renotificar y contrastar algunas posibles características genéticas polimórficas entre ambos grupos, que pudieran explicar las diferencias en la evolución de la enfermedad.
Agradecimientos. El autor agradece a Itzel Calleja, Augusto Rojas, Rocío Ortiz, Ulrich Bernard, Carmen Barboza, Francisco González, Patricia Pérez, Esthela Sandoval, Socorro Rodríguez, Teresa Plascencia, Lezmes Valdéz, Sofía Bernal y Oscar Fajardo por su colaboración y apoyo.
Fernando Lizcano Losada
Director del Laboratorio de Biología Molecular
Facultad de Medicina de la Universidad de La Sabana
Correspondencia: fernando.lizcano@unisabana.edu.co
Palabras claves: tumores hipofisiarios, genes supresores de tumores, factores de crecimiento, proliferación celular.
Resumen
Los tumores hipofisiarios son usualmente una patología benigna diagnosticada por sus diversas manifestaciones clínicas que ocasionan cambios fenotípicos en el individuo que son generados por las hormonas secretadas. Hasta el momento no se ha descifrado sí la etiología de estos tumores es consecuencia de una hiperestimulación neurohormonal hipotalámica o debido a un defecto intrínseco en la hipófisis o una combinación de ambos.
Alteraciones de factores de crecimiento debido a una hiperestimulación hormonal o la metilación del promotor de genes supresores que impiden el debido control del ciclo celular de los diferentes grupos celulares son las causas más plausibles de los adenomas hipofisiarios. Aunque varios modelos en ratones han validado algunas de las alteraciones en genes candidatos como los tumores supresores MEN1, pRB o p53 y la activación de oncogenes como Ras, no se ha observado una evidencia clara que estos factores estén involucrados en los humanos. Las mutaciones genéticas que están involucradas en otras neoplasias son raramente observadas en los tumores de hipófisis y solamente una pequeña cantidad de tumores hipofisiarios son originados por desórdenes heredados como la neoplasia endocrina múltiple 1 ( MEN1). Es posible que cada grupo celular sea productor de hormona de crecimiento, prolactina, gonadotropinas ó corticotropina tenga una alteración particular en la proliferación celular que ocasiona la expansión clonal del grupo celular específico.
Helena Groot y María Mercedes Torres
Laboratorio de Genética Humana – Universidad de Los Andes
Correspondencia: hgroot@uniandes.edu.co
Palabras claves: Cáncer gástrico, genes detoxificantes, polimorfismos, RFLP-PCR
Resumen
El cáncer gástrico (CG) es una de las principales causas de mortalidad por cáncer en Colombia. Es una enfermedad de etiología compleja, que involucra factores genéticos y ambientales como: la dieta, agentes infecciosos (H.pylori), y exposición ocupacional. Así como factores genéticos del huésped; polimorfismos en enzimas encargadas de la desintoxificación de xenobióticos (glutationes, citocromo p450), enzimas involucradas en la reparación de ADN (XRCC1), de proteger la mucosa gástrica (MUC1) y factores inmunogenéticos (IL1B y FNT) se han relacionado con la susceptibilidad a CG.
La incidencia de CG a nivel mundial es variable y ha sido atribuido a la distribución diferencial de estos polimorfismos en las poblaciones. El propósito de este estudio fue evaluar la asociación de factores ambientales y genéticos con CG en dos poblaciones colombianas (una étnicamente heterogénea (Bogotá I) y otra étnicamente homogénea (Cauca II). La delección homocigótica de las enzimas glutation S-transferasas M1 (GSTM1) y T1 (GSTT1) fueron determinadas por PCR y por RFLP-PCR las variantes genéticas de las enzimas CYP2E1 y XRCC1.
En la población I se analizaron 68 pacientes con CG y 111 donantes sin CG y en la población II 46 pacientes con CG y 96 sin CG. La asociación entre los polimorfismos y los factores ambientales con CG fueron evaluados mediante análisis de regresión logística. Se observó asociación entre las variantes genéticas del gen XRCC1 (Arg/Arg 194 ) y CG en la población I (OR:4.15; CI 95% [1.33 -12.9]). El habito de fumar y el consumo de comidas ahumadas estuvo asociado con CG (OR:3.96; CI 95% [1.61-2.81]) y OR:2.75; CI 95% [1.29- 5.87], respectivamente). En la población II, el polimorfismo de delección de la enzima GSTM1 fue asociado con CG (OR:5.45; CI 95% [1.72- 17.20]).
Algunos factores como fumar, consumo de alcohol e infección con H.pylori mostraron asociación con CG (OR: 6.70; CI 95% [2.20-2.30]) (OR:3.27; CI 95% [1.14-9.4]) (OR:5.58; CI 95% 1.81-17.19]). La diversidad étnica en estas poblaciones podría ser la causa de la variación en las frecuencias génicas de estos marcadores de susceptibilidad a cáncer y explicar parcialmente los resultados encontrados en estas dos poblaciones.
El hábito de fumar es un importante factor de riesgo para CG en regiones de alta incidencia en Colombia.
Dr. Hugo A. Barrera Saldaña1 y Dr. Eduardo Aguirre P2.
1Facultad de Medicina de la Universidad Autónoma de Nuevo León. Av. Madero
2S/N, Col. Mitras Centro, C.P. 64460, Monterrey, Nuevo León. México.
Correspondencia: hbarrera@fm.uanl.mx
Palabras clave: genómica, diagnóstico molecular, familias multigénicas, biotecnología.
Resumen
En Abril del 2003 se concluyó el Proyecto del Genoma Humano, numerosos avances y descubrimientos fueron piezas claves para agilizar la elucidación de la enciclopedia hereditaria de nuestra especie. La secuenciación manual del locus hGH, el mismo que alberga los genes de las hormonas del crecimiento (GHs) y somatomamotropina coriónica (CSHs), también conocida como lactógeno placentario. La información genética generada (~66,500 nucleótidos), guió en nuestros laboratorios los experimentos que suscitaron los descubrimientos e invenciones descritos en forma resumida a continuación.
Con el uso de iniciadores consenso y la Reacción en Cadena de la Polimerasa (PCR), se obtuvieron, clonaron y secuenciaron los genes y sus regiones intergénicas (sitio donde se encuentra el promotor). La secuencia del locus en cada especie estudiada, sirvió de base para determinar el patrón evolutivo de la familia GH en primates. En el caso de los genes, éstos se transfectaron en células en cultivo para caracterizar la expresión, identidad bioquímica e inmunológica del producto génico, mientras que para el caso de los promotores se les unió el gen reportero ß-Gal para evaluar su potencia y especificidad tisular; por otro lado estos mismos promotores se clonaron en vectores adenovirales regulando la replicación de los mismos de manera tejido-específica. También, el ADNc producto de cada gen del locus humano fue introducido en el genoma de la levadura Pichia pastori, para producir y caracterizar las proteínas recombinantes de interés. Posteriormente, se procedió a semipurificar estas últimas para comparar sus actividades biológicas. Confirmado el éxito de la metodología en el locus humano, ésta se extrapoló a otras especies de mamíferos, los logros de traducir el conocimiento genómico del locus hGH/CSH fueron:
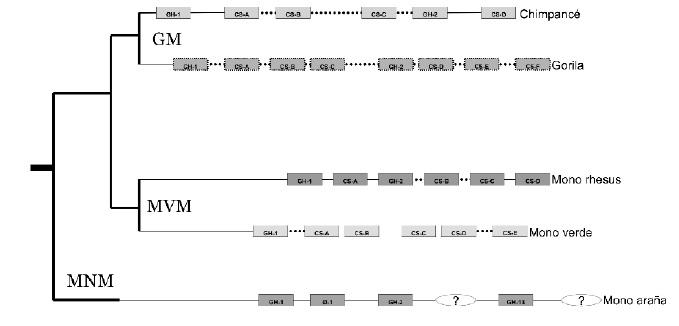
a) El aislamiento de genes y regiones intergénicas de loci GH de monos del Nuevo Mundo (MNM), monos del Viejo Mundo (MVM) y grandes monos (GM), para entender mejor la evolución de esta familia génica, Figura 1.

Figura 1. Cladograma y progreso de secuenciación del loci GH/CSH de diferentes especies en estudio. Cuadros y líneas sólidas representan regiones secuenciadas, los punteados a secuenciación en progreso y los vacíos a regiones aún no clonadas. GM, grandes monos; MVM, monos del viejo mundo, y MNM, monos del nuevo mundo.
b) La identificación en los promotores largos de los principales elementos de control de la especificidad tisular de expresión de los genes, Figura 2.
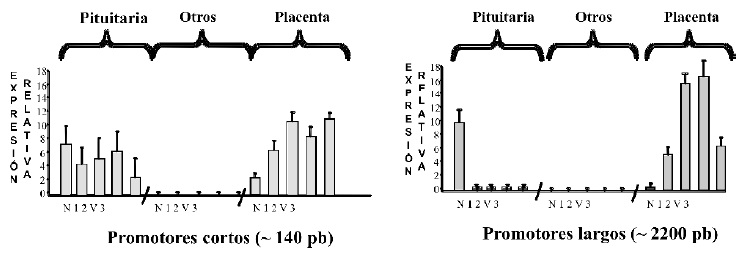
Figura 2. Descripción de los elementos reguladores de la expresión tejido específico. Ensayos de transfección de dos versiones de los promotores en líneas celulares utilizando el gen reportero b-Gal.
c) Un nuevo método para diagnosticar deleciones de los genes GHs y CSHs en pacientes en deficiencias de dichas hormonas.
d) La producción en levaduras de las proteínas codificadas por los genes miembros de la familia humana, así como de varios animales, como el gato, perro, vaca y cabra, Figura 3.
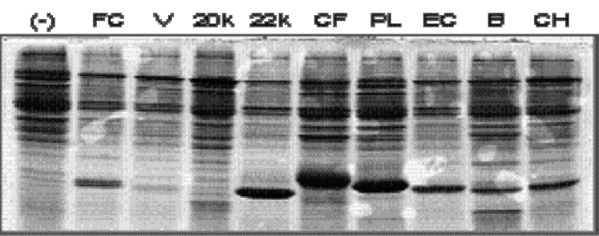
Figura 3. Ejemplo de GHs expresadas en Pichia pastoris. Proteínas en medio de cultivo resueltas en SDS-PAGE al 15%. (-) clona no inducida; resto, GHs diversas. FC, gato; V, variante; 20K y 22 K, hipofisiarias; CF, perro; PL, lactógeno; EC, caballo; B, bovino; y CH, cabra.
e) Desarrollo de nuevos vectores para terapia génica para cáncer con selectividad para tumores hipofisiarios.
Como conclusión, la regulación de la expresión génica y la dramática evolución en primates, entre otras características, hacen del locus GH/CSH un modelo excepcional para estudiar la aparición de la especificidad tisular de expresión y de hormonas nuevas durante la evolución. La disponibilidad de sus versiones recombinantes facilitará estudios sobre la evolución de las funciones de estos importantes reguladores metabólicos.
Agradecimientos. Este trabajo fue apoyado por el proyecto 25579M del CONACYT. El autor agradece apoyos de Rafael González, Agnés Revol, Iram Rodríguez, Herminia Martínez, Hipólito Castillo y Jorge Ascacio.
Martha Patricia Alba S, Adriana Bermudez, Gladys Cifuentes, Luz Mary Salazar, Fabiola Espejo y Manuel Elkin Patarroyo.
Fundación Instituto de Inmunología de Colombia
Correspondencia: patricia_alba@fidic.org.co
Palabras claves: RMN, COSY, TOCSY, NOESY, péptidos, cálculo de estructura
La espectroscopia de Resonancia Magnética Nuclear (RMN) actualmente es una de las técnicas más utilizadas para la elucidación estructural de compuestos orgánicos como lípidos, metabolitos secundarios obtenidos a partir de productos naturales, compuestos órgano-metálicos entre otros, y macromoléculas como péptidos, proteínas, oligosacaridos, fragmentos de DNA y RNA etc. Cada vez encuentra más aplicaciones en la medicina donde también es llamada RMN de Imágenes y es usada como diagnostico en diferentes especialidades (cardiología, neurología, ortopedia etc.) La RMN de péptidos y proteínas presenta dos ventajas importantes, la primera es que además de determinar el tipo de estructura presente en la molécula indica los residuos que están formando parte de dicha estructura; la segunda ventaja es que permite definir la estructura de las moléculas en solución, lo cual es importante ya que podemos determinar la estructura 3D de las biomoléculas en el mismo medio en el cual realizan su función.
La RMN se basa en la propiedad que presentan ciertos núcleos atómicos de absorber selectivamente energía de radiofrecuencia cuando se colocan bajo un campo magnético; entre estos núcleos se encuentra el 1H y 13C de gran importancia en el estudio de moléculas biológicas. Las muestras se preparan en solución de trifluoroetanol (TFE 30%, estudios con diferentes solventes han demostrado que el TFE en mezcla con agua, baja la constante dieléctrica del agua y permite que en la solución se favorezcan las fuerzas hidrofóbicas responsables de la estructura tridimensional de la molécula).
Como se mencionó anteriormente la resonancia magnética nuclear es la única técnica que puede determinar la estructura de proteínas en solución, por lo tanto su estudio por RMN puede conducir al entendimiento de la estructura y dinámica de proteínas en solución y la relación de estas propiedades con la función biológica.
Por la elevada complejidad y el gran solapamiento de señales que pueden llegar a presentar los espectros de una biomolécula, se utilizan habitualmente experimentos de RMN de dos (2D) o más (tres, 3D y cuatro, 4D) dimensiones. En el caso de moléculas de peso molecular menor de 15 kDa, la estructura puede obtenerse a partir del análisis de espectros 2D RMN homonucleares de H. Para moléculas de peso molecular hasta
30kDa es preciso aplicar métodos heteronucleares para resolver la asignación de las señales de interés. Entre los experimentos mas utilizados en RMN de 2 dimensiones se encuentran:
- El experimento COSY H-H (Correlation SpectroscopY), el cual permite obtener las interacciones que tienen lugar entre núcleos cercanos a máximo tres enlaces covalentes de distancia, para asignar los sistemas de .spin. de átomos que estén acoplados.
- El experimento TOCSY (Total Correlation SpectroscopY) permite obtener las interacciones entre protones comunicados a través de una cadena hidrocarbonada (cadena lateral), de tal manera que es posible identificar el sistema de .spin. de todos y cada uno de los aminoácidos que forman parte de la cadena peptidica.
- El experimento NOESY (Nuclear Overhauser Enhancement SpectrocopY) puede detectar interacciones entre núcleos que aunque estén lejos en la secuencia primaria, se encuentren a una distancia en el espacio menor de 5 Å.
Las estrategias de asignación espectral están bien establecidas y son distintas si se utilizan espectros homonucleares o heteronucleares. Fundamentalmente se basan en dos aproximaciones, asignaciones a partir de los efectos NOE o a partir de las constantes de acoplamientos internucleares. Una vez asignados los espectros, el siguiente paso es la obtención de las restricciones estructurales que servirán de base para el cálculo del modelo estructural con la ayuda de técnicas computacionales como el programa INSIGHT II el cual permite construir el péptido al cual se le aplicarán restricciones en las distancias hidrógeno – hidrógeno de acuerdo a los datos de distancia de los NOE, restricciones en las distancias hidrógeno - oxígeno de acuerdo a los datos de desplazamiento químico a diferentes temperaturas y restricciones de ángulo diedral Phi, de acuerdo con los datos de constantes de acoplamiento, al modelo así creado con el programa Distance Geometry se le refina mediante utilización de programas de Minimización y Dinámica Molecular implementados en DISCOVER. De este proceso se generan cincuenta estructuras de las cuales se escoge una subfamilia de estructuras que se ajusta a los datos experimentales, al final de esta subfamilia se escoge una estructura consenso que representa a la estructura tridimensional de la molécula en solución.
Caracterizando estructuralmente los péptidos (péptidos perteneciente a proteínas de malaria) por técnicas de Resonancia Magnética Nuclear hasta llegar a la obtención del modelo tridimensional a través de la metodología del cálculo de estructura, y teniendo resultados inmunológicos, el siguiente paso es establecer una posible relación estructura-actividad inmunológica.
Como resultado de esta búsqueda se ha encontrado que existen diferencias en la conformación molecular de los péptidos protectores contra la malaria respecto a aquellos que son no protectores. Estos cambios pueden involucrar que fragmentos de a hélice se presenten en diferente región y en diferente extensión del péptido ó que se registren giros de estructura b de diferente tipo en los péptidos protectores y no protectores.
Leonardo R. Lareo
Pontificia Universidad Javeriana, Facultad de Ciencias, Departamento de Nutrición y Bioquímica
Bioquímica Computacional y Estructural y Bioinformática
Correspondencia: l.lareo@javeriana.edu.co
Palabras clave: Modelos, identificación de patrones, paradigmas de investigación, secuencias, actividad química, función biológica
Resumen
Una de las mayores dificultades en la investigación interdisciplinaria, entre las ciencias biológicas y las otras ciencias naturales, radica en las diferencias entre sus paradigmas de investigación y en el desconocimiento que los científicos de cada disciplina tienen de esos fundamentos de la investigación de las otras disciplinas. En este trabajo se pretende iniciar un proceso para subsanar la brecha conceptual entre las ciencias físicas y químicas, matemáticas e ingenierías con las ciencias biológicas. Se presenta el paradigma básico de la investigación en las biociencias y algunos de los puntos integradores entre estas y las otras ciencias naturales. Se intentan ilustrar preguntas como: ¿Cuál es el paradigma básico de investigación científica en biociencias que las diferencia de las otras ciencias básicas? ¿Qué es un patrón biológico? ¿Qué es un indicador de actividad y función biológica o bioquímica? ¿Qué tipos de patrones son posibles identificar en el biomoléculas?
Introducción
La metodología fundamental para la generación de nuevo conocimiento en las ciencias biológicas es la comparación. Con el propósito de precisar el origen de esta forma o paradigma de investigación consideremos, por ejemplo, una célula viva es un ejemplo de sistema biológico. Esta tiene una frontera constituida por su membrana plasmática, internamente puede tener subsistemas, como cada una de sus organelas; núcleo, mitocondria, retículo, etc. Las variables de entrada pueden ser seleccionadas desde una amplia gama de posibilidades entre las que se encuentran las concentraciones externas de sustancias que pueden fluir hacia el interior de la misma, la temperatura del medio, la presión, etc. Las variables de salida o respuestas pueden incluir, de nuevo, concentraciones internas de sustancias, tasas metabólicas, respuestas eléctricas y mecánicas, etc. Se reconoce que el sistema será dinámico cuando el sistema presenta variaciones en el tiempo.
Estos sistemas dinámicos serán la característica fundamental de los sistemas biológicos vivos, esto solo introduce a la definición operacional el hecho de que varía temporalmente en función de las entradas y salidas.
Adicionalmente todos los sistemas, en particular los biológicos que nos ocupan ahora, tienen sistemas de control. Se reconoce una entrada de referencia, que es la entrada que genera un comportamiento o una respuesta de magnitud y sentido conocida. Toda salida del sistema dinámico esta controlada por un determinado sistema de control, que compara esta respuesta con el comportamiento generado por la respuesta a la señal de referencia. Si existe un error o desigualdad entre la respuesta real y la deseable ante una señal de entrada determinada, el sistema de control produce un elemento actuante o factor regulador que actúa sobre el sistema forzándolo a alcanzar el equivalente al producido por la señal de referencia y en esa forma reducir el efecto del error. Por ejemplo, una célula expuesta a un entorno hiperosmótico regenerará un comparación del flujo de sustancias entre el interior y el exterior hasta llegar a que se requiere dejar salir cierta cantidad de solvente tratando de reducir la osmolaridad externa y así minimizar el efecto nocivo, en lo posible, de esa señal por encima de la referencia como es tener un entorno isoomolar con el interior. El hecho de comprender este proceso no significa que se conozcan los detalles del mecanismo de regulación y control que se activan.
Es de relevante importancia precisar desde ya que todo el sistema vivo que se considere lo esta porque tiene precisos sistemas de control, denominados sistemas homeostáticos, los cuales no hacen más que realizar comparaciones entre unas señales y unas salidas con otras obtenidas en diferentes condiciones. Es decir los sistemas de regulación y control que permiten los comportamientos que caracterizan a los seres vivos se fundamentan en la comparación.
Desarrollo
Con el anterior razonamiento se llega entonces a que la comparación como paradigma de investigación en las ciencias biológicas se fundamenta en el mecanismo mismo que fundamento los procesos vitales. Los sistemas biológicos viven en función de comparaciones de señales y respuestas y la investigación se hace en términos de comparar las nuevas respuestas con respuestas previamente conocidas.
En esencia las comparaciones permiten la identificación de patrones en los sistemas biológicos pero ahora nos centraremos en el biomoléculas. Para estas, existen por lo menos, cinco tipos de patrones: los patrones de secuencia/composición; los patrones de estructura; los patrones de perfiles/valores energéticos; los patrones de actividad (química); los patrones de función (biológica).
Para la identificación de patrones de los tres primeros tipos citados se han desarrollado muchos sistemas y mecanismos, esencialmente desde la Biología Molecular Computacional y otras disciplinas, para los dos últimos los patrones de actividad y de función, dada la complejidad de los proceso biológicos, representados en cada una de las actividades de sus componentes no existe una forma de realizar mediciones de los procesos completos y toda la teoría de mediciones se realiza sobre indicadores. Este es otro importante punto a precisar, un indicador de una variable o de un proceso es otra variable, directa o derivada, que cambia en la misma dirección y sentido del evento que se quiere medir.
Las relaciones entre los patrones e indicadores identificados generan las áreas de mayor investigación como son la relación estructura-actividad, base de la farmacología moderna, la relación de la estructura-función, base de la bioquímica estructural y enzimología, la relación de estructura -secuencia, base de los algoritmos de predicción de estructura moleculares.
Conclusión
Conocer y ser conscientes de la fundamentación de nuestros paradigmas de investigación en las ciencias biológicas nos permitirá, primero, realizar más y mejores avances en nuestras disciplinas y a su tan importante como lo anterior, interactuar mejor con otras disciplinas científicas que como la física, química y matemáticas y las ciencias aplicadas, como las ingenierías, que tienen otros paradigmas de investigación.
Beatriz Restrepo C Esp Msc., Patricia Landazury MSc PhD.
Laboratorio de Bioquímica y Genética, Programa de Medicina. Facultad Ciencias de la Salud. Universidad del Quindío.
Correspondencia: beatrizrc@uniquindio.edu.co
Resumen
Los trastornos que afectan la estructura, composición o metabolismo de las lipoproteínas reciben el nombre de dislipemias o dislipidemias y se suelen agrupar en hiperlipoproteinemias, hipolipoproteinemias y aparición de lipoproteínas anómalas.
La determinación cuantitativa clásica de fracciones lipídicas en plasma, conjuntamente con la determinación de las fracciones lipoproteicas y de cada una de las apoproteínas constituyen importantes marcadores de valor diagnóstico. La determinación del tamaño de las lipoproteínas y de la relación de lípidos/apoproteínas, así como de la actividad de las enzimas implicadas en el metabolismo de las lipoproteínas, contribuyen a un diagnóstico más sensible de algunas dislipidemias. Por otra parte, la utilización de técnicas moleculares que incluyen el aislamiento de DNA, la determinación de polimorfismos génicos, la secuenciación de segmentos génicos de apoproteínas amplificados por PCR y la valoración de las alteraciones en los procesos
postraduccionales contribuyen a un mejor diagnóstico y tratamiento de las dislipidemias.
Palabras claves: dislipidemias, hiperlipoproteimenias, lipoproteínas LDL, apoproteinas B y E.
Carmen Cecilia Almonacid Urrego
Universidad Colegio Mayor de Cundinamarca
Correspondencia: calmonacid@unicolmayor.edu.co
Palabras clave: troponina, infarto agudo al miocardio (IAM), síndrome coronario agudo (SCA), mioglobina, péptido natriurético ventricular (BNP), homocisteina, Proteína C reactiva (PCR).
Resumen
De los cada 6 millones de pacientes que ingresan con dolor torácico a las salas de urgencias de los Estados Unidos, dos millones aproximadamente padecen síndrome coronario agudo (SCA), incluyendo el infarto agudo del miocardio (IAM) y la angina inestable. El infarto agudo al miocardio (IAM) presenta una tasa de mortalidad mundial de 44/100.000 habitantes y es responsable de 500.000 muertes anuales en los Estados Unidos. El diagnóstico de IAM es uno de los puntos críticos dentro de la práctica médica debido al riesgo médico legal que representa un paciente dado de alta por error y a los altos costos que genera para el sistema de salud el subdiagnóstico del síndrome coronario agudo. Por todo lo anterior, se hizo necesario encontrar nuevos métodos que permitieran detectar a tiempo y con mayor porcentaje de confiabilidad, la presencia de cualquier alteración cardiaca por pequeña que esta fuera. Es así como en este momento contamos con biomarcadores como las troponinas, la mioglobina y la homocisteina, entre otros, que facilitan la detección y la prevención de la enfermedad cardiaca.
Biomarcadores ¿Qué tenemos?
Troponina. Es un complejo de 78 KD ubicado sobre el filamento delgado de los músculos. Se encuentra conformado por las isoformas I (TnI), C (TnC) y T (TnT). Las troponinas T e I son utilizadas como marcadoras de función cardiaca.
La Troponina I, la isoforma cardiaca (TnIc), es específica para corazón, exhibe una sensibilidad similar pero una especificidad más significativa (100%) que otros marcadores de daño miocárdico. Se empieza a detectar en suero dentro de las primeras 4 a 6 horas luego del dolor, alcanza un pico máximo entre las 12 y 24 horas y regresa a la normalidad después de 5 a 9 días del evento isquémico.
La troponina T, tiene un peso molecular que oscila entre 37 y 39 KD. Tiene 2 isoformas reconocidas (muscular y esquelética), las cuales son codificadas por un mismo gen. Se detecta a las 3 horas de ocurrido el daño, alcanza su pico entre las 24 y 48 horas y puede permanecer elevada hasta el día 14 posterior a la lesión.
Mioglobina. Es una proteína muscular que presenta bastante homología con la hemoglobina del adulto.
Sus niveles se incrementan en sangre periférica de 1-3 horas después de haberse iniciado el dolor precordial, alcanzan su pico máximo entre las 6 y 9 horas y se normalizan dentro de las 24 horas.
Creatin quinasa isoenzima MB (CK MB).Corresponde a la isoenzima cardiaca de la creatin quinasa. Se incrementa entre las 4 y 6 horas después del infarto, alcanza su pico máximo entre las 12 y 24 horas y se normaliza entre las 36 y 48 horas.
Aunque por muchos años se consideró a la CK MB como el gold estándar para el diagnóstico de IAM, en la actualidad se sabe que no es específica de músculo cardiaco y se encuentra presente en un 1-3% de músculo esquelético. Péptido natriurético tipo B (BNP). Pertenece a la familia de las hormonas péptidas natriuréticas, involucradas en la regulación del sodio y el agua durante la homeostasis. Se conoce también como péptido natriurético cerebral y/o ventricular. Es secretado por los ventrículos en un 60-80% y en menor proporción por el cerebro, debido a que se libera ante un incremento en la presiones de llenado ventricular, se constituye en un marcador específico para el diagnóstico de insuficiencia cardiaca (IC) y la evaluación y diagnóstico de falla cardiaca crónica (FCC). De igual manera, se ha demostrado su utilidad como prueba de tamizaje en hipertrofia ventricular izquierda (HVI) y en la diferenciación de falla cardiaca congestiva en pacientes con disnea aguda.
Homocisteina. Es un aminoácido con un grupo sulfihidrilo libre que se sintetiza en el organismo como producto de la demetilación de la metionina, posteriormente puede transulfurarse y excretarse por la orina en forma de cisteina, o remetilarse nuevamente a metionina. los niveles séricos de homocisteina se incrementan en la fase aguda de un infarto agudo al miocardio (IAM) o un accidente cerebrovascular (ACV). Se recomienda cuantificar homocisteina en individuos con historia clínica de enfermedad ateroesclerótica y en pacientes con manifestaciones previas de enfermedad cardiaca.
Proteína C reactiva (ultrasensible). La proteína C reactiva es una globulina con una masa molecular de aproximadamente 11800 daltons, compuesta de 5 subunidades globulares cíclicas. Puede elevarse en plasma o suero en procesos infecciosos y condiciones inflamatorias como artritis reumatoide, enfermedad cardiovascular y enfermedad vascular periférica.
En prevención primaria la PCR es útil como predictor de futuros eventos cardiovasculares. Se ha encontrado que la PCR aporta información pronóstica en cada uno de los niveles de riesgo cardiovascular. En prevención secundaria su utilidad es menos certera, en el caso de un evento coronario agudo y angina inestable, predice mortalidad temprana y tardía en isquemia aguda y agrega valor predictivo a la troponina. En pacientes con dolor toráxico y niveles de troponina negativos, una PCR elevada está asociada con un incremento del riesgo a corto y largo plazo y exige modalidades adicionales de evaluación.
Biomarcadores ¿Hacia dónde vamos?
Subperfiles de LDL. Las lipoproteínas son estructuras complejas conformadas por proteínas y diferentes tipos de lípidos. Conocer cómo y cuando cambia su composición, es clave para la correcta interpretación de los subperfiles.
La composición de las LDL puede variar de acuerdo con el tamaño de las partículas y el estado metabólico de cada persona. Con respecto a la concentración de colesterol libre, se ha observado que las personas con LDL pequeño tienen menos colesterol que las personas con LDL grande. De igual manera, la composición de las moléculas de LDL puede variar de persona a persona. Es así como personas con el mismo tamaño de molécula, pueden tener partículas con más colesterol y menos triglicéridos dentro de ellas y personas con número idéntico de partículas de LDL, pueden presentar diferencias de hasta 50 mg/dl en sus niveles de colesterol LDL.
Debido al interés creciente en estas pruebas, están disponibles en el mercado diversas metodologías como RMN, ultracentrifugación, electroforesis en gel y precipitación, que permiten valorar diferentes propiedades de las partículas de LDL.
Albúmina modificada por la isquemia (AMI). Permite detectar la presencia de isquemia (paso previo al IAM), en pacientes con dolor de pecho sugestivo de origen cardiaco. Presenta como desventaja su baja capacidad para distinguir entre individuos con isquemia que desarrollarán infarto y los que no.
Adiponectina. La adiponectina es una hormona producida y secretada por los adipocitos. Sus niveles son más altos en mujeres (+/- 28 ug/ml) que en hombres (+/- 16.5 ug/ml) y se disminuyen en obesidad y diabetes tipo 2. Su utilidad clínica se encuentra en estudio, se ha comprobado que sus niveles están inversamente relacionados con la incidencia de ECV en pacientes con diabetes tipo 1; entre mayor sea el nivel de adiponectina, existe menor posibilidad de ECV en el paciente. Por lo tanto, podría llegar a ser un predictor de riesgo cardiovascular y un agente potencial para la prevención entre individuos de riesgo alto.
Patricia Landazuri MSc, PhD, Guillermo García Zuluaga.
Laboratorio de Bioquímica y Genética, Programa de Medicina. Facultad Ciencias de la Salud. Universidad del Quindío. Armenia Colombia.
Correspondencia: plandazu@uniquindio.edu.co
Resumen
Los lípidos representa entre el 30-40% de la ingesta diaria en la población occidental, dado su carácter hidrofóbico, estos lípidos de la dieta y los de origen endógeno para ser utilizados por el organismo son transportados en el plasma asociados a partículas hidrofílicas que se constituyen en macromoléculas llamadas lipoproteínas. En el organismo estas superestructuras de lípidos y proteínas están organizadas y diseñadas para intercambiar entre ellas y con las células, ácidos grasos, colesterol, esteres de colesterol y las apoproteínas.
En el metabolismo normal de las lipoproteínas, la síntesis, el intercambio de materiales y su degradación esta sincronizado y regulado por hormonas, enzimas y receptores de manera tal que se mantiene un balance estricto en esta actividades. Sin embargo, este balance se puede perder por múltiples razones, entre ellas las genéticas (polimorfismos, delecciones rearreglos) y las de estilo de vida (dieta, sedentarismo, consumo de alcohol, cigarrillo entre otros). Este cambio en el metabolismo normal con lleva a alteraciones fisiológicas que se manifiestan con patologías comunes y muy frecuentes en nuestra población como la arterosclerosis. La investigación reciente ha aumentado nuestros conocimientos sobre el metabolismo de las lipoproteínas y su papel en la aterogénesis, recientemente la investigación en lipoproteínas se ha dirigido hacia el metabolismo de las HDL con el objetivo de identificar nuevos blancos terapéuticos en el tratamiento aterogénico.
Palabras claves: Enfermedad vascular, hiperlipoproteinemias, lipoproteínas HDL, CETP