Influencia del a-Factor sobre la expresión de IDShr en Pichia pastoris: Revisión sistemática de literatura y análisis computacional
Carlos E. Barragán1, Homero Sáenz1,2 Raúl A. Poutou1,3, Henry A. Córdoba1,4, Marcela M. Mercado5, Luis A. Barrera1
1 Instituto de Errores Innatos del Metabolismo,
2Decanato de Medicina, Universidad Centroccidental Lisandro Alvarado,Barquisimeto, Venezuela.
3 Grupo de Biotecnología Ambiental e Industrial, Departamento de Microbiología.
4Departamento de Química.
5 Grupo de Enfermedades Infecciosas, Departamento de Microbiología, Pontificia Universidad Javeriana, Bogotá, Colombia.
Correspondencia: abarrera@javeriana.edu.co , mmercado@javeriana.edu.co
Recibido: 04-07-05 / Aceptado:08-08-05
Palabras clave: revisión sistemática, péptido señal, expresión de proteínas, Pichia pastoris
Key Words: systematic review, signal peptide, protein expression, Pichia pastoris
Una de las alternativas propuestas en los últimos años, es la terapia de reemplazo enzimático (TRE), la cual ha mostrado la posibilidad de revertir las complicaciones viscerales en enfermedades severas como el Síndrome de Hunter (6). El objetivo de la TRE es proporcionarle al paciente la enzima purificada de origen exógeno, con la intención de que ésta llegue a los tejidos en los cuales está ausente, lo que podría ser una opción para el tratamiento de la MPS II, siempre y cuando se logre obtener cantidades suficientes de la enzima, no antigénica y con alta actividad específica (7). Para obtener cantidades clínicamente útiles, la enzima puede ser sintetizada en modelos biológicos recombinantes como bacterias, levaduras o líneas celulares de mamíferos. Uno de los sistemas de expresión que ha mostrado ser eficiente para la expresión de proteínas glicosiladas como la IDSh es la levadura metilotrófica Pichia pastoris (8).
P. pastoris ha sido utilizada como modelo biológico para la expresión extracelular de diversas proteínas humanas, entre otras la a-galactosidasa A (9), la cistatina C (10), la glucocerebrosidasa (11) y la angiostatina (12) y en nuestro grupo la Iduronato Sulfatasa (13). Esta levadura ha sido utilizada porque ofrece ventajas con respecto a otros sistemas biológicos. Entre los principales aspectos, están los altos niveles de expresión de proteínas como consecuencia de las densidades celulares que se pueden obtener en cultivo, la facilidad para implementar la producción en bioreactores a gran escala y, la estabilidad genética.
La mayor ventaja con relación a los sistemas procarióticos, es la capacidad de llevar a cabo modificaciones post-traduccionales bastante similares a los organismos eucarióticos superiores. Entre las modificaciones se encuentra el procesamiento de las secuencias señal, el plegamiento de las proteínas, la formación de puentes disulfuro y las N y Oglicosilaciones, sin generar hiperglicosilación a diferencia de lo que sucede en S. cerevisiae (14-17).
La expresión de proteínas heterólogas en P. pastoris se ha logrado gracias a la utilización del promotor AOX1, que es reprimido fuertemente cuando las células crecen sobre glucosa y otras fuentes de carbono (represión/ derepresión por catabolito), e inducido en proporción ~1000:1 cuando crecen en medio suplementado con metanol como única fuente de carbono (inducción específica por sustrato) (14, 15).
Por lo general, se ha optado por este sistema para la secreción de proteínas recombinantes, debido a que la recuperación de la molécula del medio de cultivo representa un primer paso en el proceso de purificación (18). Con el fin de obtener una secreción eficiente el ADNc se fusiona con una secuencia líder compatible por el modelo de expresión, lo que además, favorece la estabilidad del sistema (19); la señal de secreción empleada con mayor frecuencia en P. pastoris, es el péptido a-Factor (a-MF) de S. cerevisiae (15), que facilita el transporte de la proteína a través del retículo endoplásmico (RE) y el complejo de Golgi, antes de su ubicación en las vesículas de secreción. La secuencia completa del a-MF consta de 85 aminoácidos (AA), los primeros 19 residuos corresponden al péptido señal y los siguientes 66 a la región denominada pro-secuencia, que contiene tres sitios potenciales de N-glicosilación en los residuos 4, 38 y 48 (20, 21).
En P. pastoris varias proteínas recombinantes han sido secretadas eficientemente, utilizando el péptido señal nativo heterólogo, mientras que otras han mostrado rendimientos dos o tres veces superiores, cuando utilizan un péptido señal de levadura (a-MF, PHO1 ó SUC2) (17). Sin embargo, hasta el momento no existen mecanismos para predecir si la señal de secreción a-MF, proporciona los mejores niveles de secreción para enzimas como la IDShr. Por esta razón, se decidió implementar una Revisión Sistemática de la Literatura, método característico de la medicina basado en la evidencia (22-24), con el propósito de recopilar información que proporcione conocimientos acerca de factores relacionados con el péptido líder y su posible incidencia sobre los niveles de secreción y expresión de proteínas humanas glicosiladas, en el modelo P. pastoris.
Búsqueda de la información.
Para la búsqueda de la información se consultaron cinco bases de datos, con el fin de recopilar las publicaciones cuyo objetivo fuera la expresión de proteínas humanas en P. pastoris: MEDLINE, Science Direct, Hinari, ProQuest (Biology y Science Journals) y SciELO. Los artículos podían estar escritos en inglés, español o portugués y publicados después de 1984 (época en la cual se empezó a utilizar P. pastoris como modelo biológico para la expresión de proteínas). Se utilizaron diferentes opciones de búsqueda empleando combinaciones de los siguientes términos: Pichia pastoris, expression proteins, extracellular proteins, pPIC9, signal peptide, secretion signal, a-Factor y a-MF. Adicionalmente se buscaron en la red los artículos referenciados que estuvieran relacionados con el tema.
Criterios de inclusión de los artículos:
a. P. pastoris como modelo biológico para la expresión de la proteína.
b. Que se hayan utilizado plásmidos como pPIC9, pPIC9K y pPICZa, que incorporan el a-Factor como señal de secreción extracelular.
c. Las moléculas heterólogas expresadas podían ser enzimas o proteínas estructurales heterólogas expresadas en P. pastoris.
d. La proteína expresada debía ser de orígen humano.
e. La proteína expresada debía presentar al menos un sitio potencial de N-glicosilación.
f. El peso molecular molecular relativo de la proteína expresada debía oscilar en el rango de 10 a 70kDa.
Los criterios de selección se definieron tratando de aproximarse a las características de la IDSh.
Evaluación de los artículos pre-seleccionados.
En este proceso se realizó una segunda selección de los estudios por medio de la valoración de su calidad, para asegurar que sólo los estudios relevantes y con un nivel aceptable fueran incluidos. Se tuvo en cuenta el nivel de impacto de las revistas donde aparecían las publicaciones seleccionadas y se verificó que el protocolo descrito en materiales y métodos estableciera detalladamente las variables de estudio tenidas en cuenta para asegurar la confiabilidad de los resultados.
Este proceso estuvo a cargo de dos asesores del trabajo, para evitar decisiones sesgadas y subjetivas.
a. En esta valoración las revisiones de literatura, notas y disertaciones fueron excluidas por no presentar datos obtenidos de estudios experimentales.
b. Sólo se emplearon las publicaciones donde se comparaba la utilización del a-Factor y el péptido señal nativo heterólogo, para la expresión de la misma proteína.
c. Fue requisito que en los resultados se incluyeran datos numéricos del nivel de expresión obtenido y/o comparaciones entre los niveles obtenidos con diferentes péptidos señales (Valor cuantitativo).
Recopilación y síntesis de datos
a. Los datos requeridos en los criterios de inclusión, se incorporaron a una base de datos de Microsoft® Access, para identificar todas las publicaciones que debían ser sometidas a la segunda evaluación.
b. De los artículos finalmente seleccionados, se obtuvieron los valores que indicaban los niveles de expresión.
Análisis estadístico.
El método estadístico empleado para evidenciar un efecto del a-MF sobre el nivel de expresión, fue la prueba de hipótesis para comparación por parejas
(.Test. t pareado) (25).
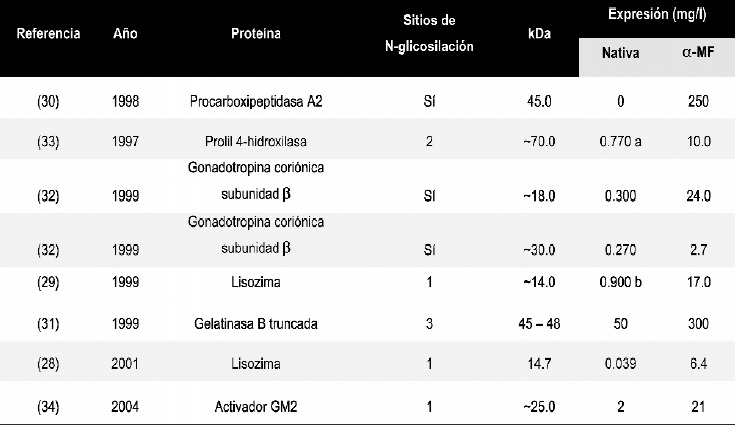
Como valores muéstrales se tuvieron en cuenta los niveles de expresión obtenidos, para las proteínas reportadas en las publicaciones incluidas en el estudio, cuando se utilizaba la señal de secreción nativa y cuando las proteínas eran secretadas con el a-MF. Para homogenizar los datos de los diferentes artículos, la información se expresó en mg/l de proteína (Tabla 1).
Tabla 1. Artículos incluidos en el estudio. a. Valor estimado a partir de la actividad enzimática, se calculó de acuerdo a la proporción de aumento que se obtuvo en la actividad de la enzima utilizando el a-MF con respecto a la señal de secreción nativa. b. Se empleó un péptido sintético similar al humano pero derivado de la lisozima de ave.

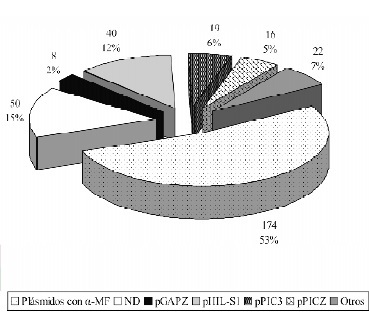
Figura 1. Clasificación de artículos teniendo en cuenta el vector de expresión utilizado, se seleccionaron aquellos que incorporaban el a-MF como señal de secreción.
Análisis computacional
Con el propósito de comparar la secuencia nativa de la IDSh y la expresada en nuestro grupo (13), se realizó el alineamiento de las secuencias, utilizando el programa BLAST de NCBI (26). La secuencia del ADNc de la IDSh nativa empleada, fue la reportada porWilson et al., (27), a la cual se accede en GenBank con el número AAA63197 y la secuencia nucleotídica que codifica para la enzima recombinante, se obtuvo a partir de la elongación del .primer. sentido, diseñado para la PCR utilizada en la comprobación de la inserción y la orientación del ADNc en el vector de expresión (13). Igualmente, se alinearon el péptido señal de la IDSh y el de S. cerevisiae (a-Factor) y se determinaron los perfiles de hidrofobicidad (ProtScale//Expasy), con el objetivo determinar una posible similaridad.
Se identificaron 349 publicaciones del total (~2000) de referencias encontradas en las diferentes bases de datos consultadas. Con la lectura de los resúmenes, se identificaron 329 artículos en los que el objetivo era la expresión de alguna proteína en P. pastoris, los otros 20 artículos se relacionaban con características genéticas de la levadura.
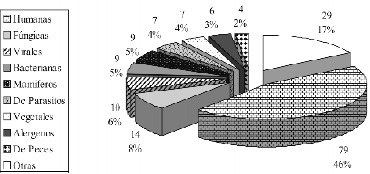
Figura 2. Selección de los artículos en los que se expresaban proteínas de origen humano del total de proteínas secretadas con el a-MF.
Los 329 artículos fueron incluidos en la base de datos de Microsoft Access®, con el fin de identificar todas las publicaciones que cumplieran los criterios de inclusión establecidos. Como se observa en la figura 1, según el plásmido utilizado para la expresión de la proteína recombinante, en 174 de los artículos (53%) se utilizaron los vectores pPIC9, pPIC9K y pPICZa, que incorporan el a-MF como señal de secreción.
Posteriormente, se seleccionaron todos aquellos artículos en los que las proteínas eran de origen humano. En la figura 2, se observa que 79 de estos artículos (46%), cumplieron con el criterio de selección.
La evaluación detallada de los 79 artículos ó referencias potencialmente relevantes, permitió identificar finalmente, 7 publicaciones (2%) en las que se comparaba el niveles de expresión obtenidos con el a-MF y el obtenido utilizando la señal de secreción nativa humana (28-34), por lo cual se incluyeron en el análisis descriptivo de la información.
Recopilación y análisis de datos
De las 7 publicaciones incluidas se extrajeron los datos más relevantes para el análisis de la información (Tabla 1). En el artículo publicado por Sen Gupta y Dighe (32), se reporta la expresión de dos subunidades diferentes de una proteína, sin embargo, por presentar diferentes pesos moleculares y por tener ambas sitios potenciales de N-glicosilación, se consideraron expresiones independientes.
El resultado de la comparación de los niveles de expresión se observa en las Figuras 3 y 4, en las que se evidenció, tanto en concentración (mg/l) como en proporción, que el a-MF en todos los casos generó mayor concentración de la proteína recombinante, en el medio de cultivo, comparado con la proteína expresada con la señal de secreción nativa heteróloga.
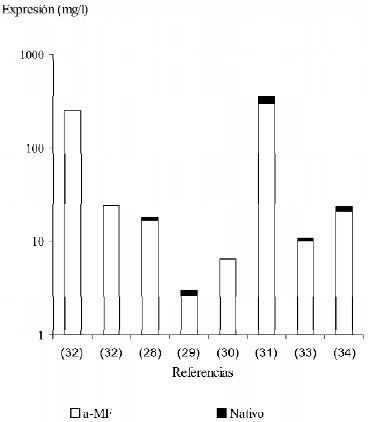
Figura 3. Comparación de los niveles de expresión en mg/l de proteína secretada al medio de cultivo, empleando diferentes señales de secreción.
El análisis estadístico se realizó con base en los datos obtenidos en los artículos seleccionados. Con este análisis, se llegó a la conclusión que existe evidencia estadística (p=0.053) para asegurar que al utilizar el a-MF como señal de secreción, se incrementan los niveles de expresión de la proteína recombinante, con un grado de significancia del 10%, de acuerdo con los datos suministrados por la literatura científica.
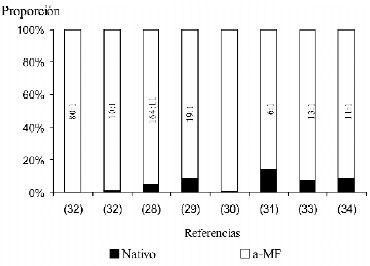
Figura 4. La proporción de cantidad expresada con el a-MF comparada con la cantidad de proteína recuperada con la señal de secreción nativa se presenta como porcentajes.
Análisis Computacional
La alineación de las secuencias y la traducción conceptual de las mismas, empleando .tools translation. de Expasy (http://www.expasy.ch ), mostró que la secuencia insertada en el plásmido de expresión contenía los 25AAdel péptido señal y los 8AAcorrespondientes a la pro-secuencia nativa de la IDSh. De otro lado, la alineación de los dos péptidos señal (a-factor vs. PS nativo de IDSh) mostró un porcentaje bajo de identidad, sin embargo a pesar de que ambos péptidos son hidrofóbicos el perfil de hidrofobicidad fue diferente (Figura 5).
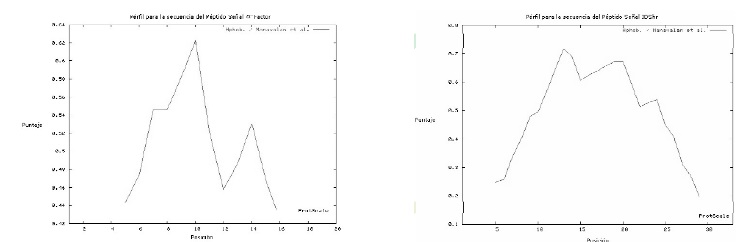
Figura 5. Perfiles de hidrofobicidad de los péptidos señal a-factor (A) y de la IDSh (B). Análisis computacional realizado en ProScale de Expasy (http://www.expasy.ch ).
Efecto del a-MF sobre el nivel de expresión
En aquellas publicaciones en las que se expresaron proteínas humanas en P. pastoris, con pesos moleculares entre 10 y 70kDA y al menos un sitio de Nglicosilación, se observó que, la señal de secreción putativa de S. cerevisiae favoreció la secreción extracelular, en comparación con los resultados obtenidos al utilizar el péptido líder heterólogo; información que fue corroborada con el análisis estadístico de los datos.
Aunque la utilización del a-MF tiende a aumentar el grado de .expresión-secreción. en todas las proteínas estudiadas, el incremento oscila en proporciones que van desde 6:1 como ocurre con la gelatinasa B (31), hasta 164:1 como ocurre con la lisozima (28). Por esta razón, no fue posible estimar el número de veces que se incrementa la expresión de una proteína humana glicosilada en P. pastoris, cuando se emplea el péptido señal de S. cerevisiae.
Teniendo en cuenta que la eficiencia del proceso de exportación de una proteína recombinante desde el RE al medio extracelular, es una etapa fundamental para el nivel de expresión, es posible que el péptido señal heterólogo de la IDSh no sea reconocido ni procesado por la levadura. Debido a que la función de la señal de secreción está directamente relacionada con el paso del polipéptido naciente a través de la membrana del RE y la interacción con las moléculas de la vía de secreción (35), es de esperar un mejor reconocimiento y procesamiento de la señal de secreción cuando ésta proviene de otra levadura.
Expresar proteínas heterólogas humanas en levaduras, utilizando el péptido señal de otras levaduras favorece el procesamiento correcto del mismo, la interacción con las proteínas auxiliares del RE para la formación de puentes disulfuro, el plegamiento y transporte por la vía de secreción (35). De no presentarse un procesamiento correcto de la proteína cuando es sintetizada, precedida de su secuencia señal nativa, podría estarse generando una proteína que termodinámicamente sea inestable y susceptible de ser empleada como fuente de aminoácidos, al ser digerida por las proteasas intracelulares. La proteólisis evidentemente afecta la recuperación de la proteína de interés en el medio extracelular.
Aunque la secuencia líder a-MF es propia de S. cerevisiae, es reconocida por las moléculas involucradas en la vía de secreción en P. pastoris; lo que indica que, las diferencias en el mecanismo de secreción de proteínas, en estas dos levaduras, es mínima. Incluso, los niveles de proteína expresada con el a-MF pueden ser superiores, si se comparan con los obtenidos utilizando el péptido señal de la fosfatasa ácida (PHO1), enzima nativa de P. pastoris, según demostraron Murasugi y Tohma-Aiba cuando expresaron midkina humana con las dos señales de secreción (36, 37).
Glicosilación y nivel de expresión.
La ventaja que ofrece el péptido líder del a-MF, sobre el péptido señal PHO1 y los péptidos nativos heterólogos, podría centrarse en los sitios potenciales de glicosilación (tres) que posee la pro-secuencia de a-MF. Gracias a los oligosacáridos unidos al péptido, en el RE se favorece el transporte de la proteína hacia el complejo de Golgi y de ahí a las vesículas de secreción. Uno de los ensayos que respaldan esta hipótesis fue realizado por Kjeldsen et al. (38); en este trabajo, se modificaron los tres sitios de N-glicosilación del a-Factor, para la expresión de una proteína recombinante en S. cerevisiae, y se encontró que las mutaciones en las secuencias Asn-X-Ser/Tre generaban reducciones drásticas en la secreción de la proteína, lo que pone en evidencia la importancia de los N-oligosacáridos en el transporte de la proteína recombinante (39, 40).
La ausencia de una pro-secuencia con diferentes sitios de N-glicosilación, podría ser un factor que ha incidido en las diferencias en los niveles de expresión obtenidos, cuando las proteínas tenían como péptido líder su señal de secreción nativa; lo anterior, podría deberse a una disminución en el reconocimiento e interacción de la vía secretoria de la levadura con el péptido foráneo. En el caso del a-MF, se pudo constatar que emplear únicamente el péptido señal sin la pro-secuencia desfavorece los niveles de expresión de la lisozima humana (29).
Peso molecular y nivel de expresión.
Según Kjeldsen et al (35), el péptido señal puede influir en la eficiencia del paso de un polipéptido de tamaño pequeño a través de la membrana del RE, debido a que el líder media el transporte rápido de la proteína en el lúmen del RE (35). Según este argumento es coherente suponer que, si la proteína no es grande (bajo peso molecular), el proceso de secreción puede ser más rápido, debido al transporte más sencillo para la célula. En el caso de los artículos incluidos en el estudio, las proteínas que presentaron los niveles de expresión más altos, tenían un peso molecular promedio de ~45kDa (30, 31) y su proceso de secreción quizás fue más sencillo para la levadura, en comparación con el caso de la subunidad a de la Prolil 4-hidroxilasa, con peso molecular de 70kDa (33). Aunque, se recomienda el uso del a-MF por presentar un efecto marcado en la secreción de proteínas de bajo peso molecular (41), proteínas con más de 70kDa, como el precursor de la proteína amiloide (APLP2), se han llegado ha expresar en concentraciones de 40mg/l (42), por lo cual este péptido líder, podría ser el más adecuado para la expresión de varios tipos de proteínas humanas, independientemente del peso molecular.
Modificaciones en la pro-secuencia del a-MF
Uno de los aspectos que caracterizaban a todas las proteínas expresadas, en los artículos estudiados, fue la eliminación de la secuencia señal nativa cuando eran expresadas con el a-MF, debido a que el ADNc que se introducía en el plásmido codificaba solamente la secuencia madura de la proteína de interés (sin péptido señal). Una de las razones por la cual se opta por sustituir el péptido líder nativo heterólogo, puede ser el efecto negativo que éste tenga sobre el procesamiento de la proteína en la levadura. Un ejemplo a considerar es el experimento desarrollado por Pham et al., (43), en el cual expresó la granzima B, y no encontró actividad al dejar la pro-secuencia de la proteína después del a-factor, mientras que la enzima expresada sin la pro-secuencia mostró actividad similar a la enzima nativa (43). Posiblemente los dos péptidos que conforman la pro-región de esta enzima, producen un efecto inhibitorio sobre su actividad, sin embargo, no se podría descartar que la pro-secuencia de una proteína afecte su actividad biológica, como consecuencia de los resultados en el procesamiento, el plegamiento y la secreción extracelular, si antes no es degradada completamente.
La estabilidad de una proteína puede verse afectada cuando no es procesada correctamente durante la secreción, debido a que cualquier segmento de aminoácidos adicional a la secuencia madura incide sobre la estabilidad termodinámica de la proteína (44).
La presencia de segmentos adicionales al péptido lí- der funcional, afecta notoriamente la eficiencia de la secreción y por tanto sobre el nivel de expresión (35). Así como se ha observado que la secuencia líder (a-Factor) presenta una estructura que facilita el reconocimiento y procesamiento en el sistema de secreción de P. pastoris, también se ha observado que el incremento en el número de residuos cargados negativamente, en el entorno al sitio de procesamiento del pro-péptido líder ó la adición de puntos de Nglicosilación adicionales, ha sido crucial para aumentar los niveles de secreción de algunas proteínas recombinantes (39).
Implicaciones del a-MF como señal de secreción para la IDShr
Teniendo en cuenta la información proporcionada en las publicaciones, siempre que se emplee el a-MF como señal de secreción, se esperaría que los niveles de expresión alcanzados con la enzima IDShr fueran superiores a los que se puedan encontrar si se utilizara la secuencia líder nativa de la IDSh. Sin embargo, a diferencia de los estudios incluidos, en los que ninguna de las proteínas secretadas con el a-MF llevaba su secuencia líder, el ADNc de la IDSh usado en el constructo pPIC9-IDShr incluye la secuencia líder nativa de la IDSh (8).
El ADNc completo de la IDSh codifica un péptido de 550 AA, de los cuales los primeros 25 constituyen el péptido señal, después de esta secuencia hay otros 8 residuos que son eliminados de la pro-proteína en el proceso de maduración (45). Como se mencionó anteriormente, la presencia de algunos residuos adicionales en la secuencia líder del a-MF pueden afectar el procesamiento, favorecer la inestabilidad de la enzima recombinante y por tanto, generar algún tipo de inestabilidad en su secreción. Además, una alteración en la secuencia del pro-péptido le impediría cumplir su función de auxiliar a la proteína, en la conformación de una estructura estable y en la protección contra la degradación. No obstante, la construcción genética pPIC9-IDShr que incluye el líder del a-factor de S. cervisiae y el péptido señal nativo de la IDSh, resultó ser funcional en P. pastoris GS115 (46) y se logró detectar actividad biológica (8, 13).
Al mantener la señal de secreción nativa la enzima puede no ser secretada eficientemente, pues se ha demostrado que cuando las proteínas sufren alteraciones en la conformación estructural no pueden ser secretadas (35). Este mecanismo de control se ha estudiado en el RE de las levaduras, especialmente en S. cerevisiae (47). Una fracción de las proteínas destinadas a la secreción es retenida y degradada, porque se restringe la exportación únicamente a las proteínas plegadas correctamente. Las proteínas plegadas incorrectamente son eventualmente llevadas al citosol para ser degradadas en el proteosoma.
Cuando una proteína no presenta grandes errores en el plegamiento, pero es termodinámicamente inestable, es transportada a las vacuolas por un receptor ubicado en las membranas de Golgi y allí son degradadas (48, 47). El cuestionamiento radica en entender si la IDShr producida P. pastoris es reconocida por el hospedero como correcta o incorrectamente plegada y si es termodinámicamente estable o no.
Para incrementar el nivel de expresión se debería eliminar la señal de secreción nativa de la IDS humana, basados en las cuatro hipótesis siguientes:
a. La proteasa señal I (E.C. 3.4.21.89), encargada del reconocimiento del a-MF, equívocamente reconoce el péptido señal de la IDSh y, al hidrolizarlo en la membrana de RE de P. pastoris, se pierde el pro-péptido del a-MF. En este caso la enzima no sería secretada por la falta de la ayuda del a- MF. Es probable que la proteína sea enviada al lisosoma debido al reconocimiento del péptido pro-
IDS (Figura 6).
b. El reconocimiento de la endoproteasa Kex2 (3.4.21.61) de P. pastoris, es el adecuado, en la pro-secuencia de a-MF; sin embargo, se podría secretar una proteína recombinate con un plegamiento inadecuado como consecuencia de la región hidrofóbica de 25 AA (PS) y la región hidrofílica de 8 AA (pro-secuencia) correspondiente al péptido líder nativo de la IDSh, secretándose por tanto una proteína probablemente inactiva.
c. La proteasa señal I encargada del reconocimiento del a-MF lo reconoce correctamente; una vez que la proteína (IDShr) se encuentra dentro del RE el plegamiento no le permite ser guiada a Golgi y por tanto podría ser degradada en el proteosoma.
d. La proteasa señal I corta correctamente en el a-MF y la enzima pasa por el RE y llega a Golgi. Una vez allí, Kex2 reconoce equívocamente la pro-secuencia nativa de la IDSh, lo que traería consigo la secreción correcta (similar a células humanas) de la proteína recombinante.
Análisis computacional
Con el objetivo de reunir evidencias para intentar esclarecer las hipótesis anteriores, se decidió comparar la secuencia de aminoácidos del a-MF y la secuencia líder de la IDSh. En este análisis se encontró que entre los primeros 11 aminoácidos de estos dos péptidos señal se presentan residuos con una alta hidrofobicidad y aunque en el resto de la secuencia la tendencia presenta más variación, estos primeros AA justificarían el corte equívoco a cargo de la peptidasa señal I de P. pastoris, si se tiene en cuenta que su acción no depende de secuencias de aminoácidos específicas, sino que está dirigida a la región más hidrofóbica del polipéptido naciente (que presenta una diferencia significativa en el punto isoeléctrico, con respecto a otros segmentos de la proteína) (49), por lo cual proporciona cierto respaldo a la primera hipótesis (a).
Analizando la secuencia nucleotídica de la IDSh, no se encontraron secuencias Lis-Arg ni Arg-Arg en el pro-péptido nativo, por lo cual es poco probable que la endopeptidasa Kex2 realice algún corte proteolítico en el segmento de 8 AA, lo que deja sin respaldo la cuarta de las hipótesis (d); favoreciendo la segunda hipótesis (b).
Es importante tener en cuenta que esta información está basada en la evidencia y representa una aproximación teórica que debe ser complementada con estudios experimentales, que corroboren si los criterios estudiados en esta revisión, pueden ser aplicados a la expresión de IDShr en P. pastoris o a otras proteínas humanas con características similares.
Teniendo en cuenta los resultados obtenidos en este estudio y con el objetivo de acercarnos teóricamente a la incertidumbre que genera la hipótesis (b), se ha comenzado el análisis computacional para modelar el efecto que se produce en el plegamiento de la IDSh, cuando está unida a los 33 aminoácidos de la secuencia señal nativa. Tratando de esclarecer si esta región (PS) altamente hidrofóbica, podría alterar en alguna medida la actividad biológica de la proteína. De otro lado, se ha comenzado el seguimiento intracelular de la proteína humana recombinate por inmunomicroscopía electrónica, utilizando un anticuerpo policlonal (IgGaIDSh) (50), para la detección de la IDShr y un anticuerpo comercial aIgG de conejo, marcado con oro coloidal (aIgG-Au).
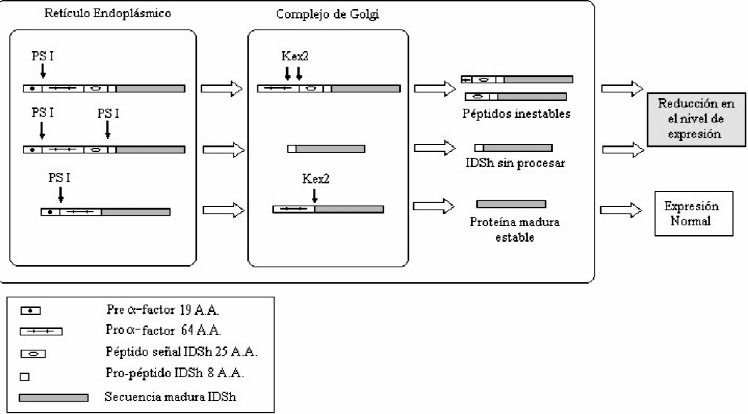
Figura 6. Posibles alteraciones de la secreción, causadas por la presencia del péptido señal nativo de la IDSh. a. Se generan péptidos inestables por la presencia de aminoácidos adicionales o por el corte aberrado de la secuencia por la enzima Kex2. b. La peptidasa señal I (PS I) realiza el corte del péptido señal en la membrana del RE y la proteína es liberada del a-Factor y queda con la pro-proteína. c. Péptido recomendado en el que se elimina completamente el péptido señal de la IDSh.
Es claro que emprender una nueva clonación del ADNc de IDSh sin el péptido señal nativo es un paso a seguir, de forma tal que pueda realizarse un estudio comparativo de la expresión en IDShr en P. pastoris con las construcciones genéticas anteriores.
De otro lado, la utilización del péptido señal de secreción a-MF de S. cerevisiae, demostró ser más útil que el homólogo (PHO1) de P. pastoris y que la secuencia líder nativa de IDSh, para la expresión de proteínas que deban ser secretadas al medio de cultivo.
Adicionalmente, se propusieron estrategias de investigación para verificar los posibles conflictos que pueden estar ocurriendo entre las secuencias líderes que resultaron incluidas en la construcción pPIC9-IDShr.
2. Albano LMJ, Sugayama SSMM, Bertola DR, Andrade CEF, Utagawa CY, Puppi F, Nader HB, Toma L, Coelho J, Leistner S, Burin M, Giugliani R, Kim CA. Clinical and Laboratorial Study of 19 Cases of Mucopolysaccharidoses. Rev Hosp Clín Fac Med Sao Paulo 2000; 55 (6): 213-18.
3. Kettlesa DI, Sheppardb M, Liebmannc RD, Davidsona C. Left Ventricular Aneurysm, Aortic Valve Disease and Coronary Narrowing in a Patient with Hunter.s Syndrome. Card Pathol 2002; 11: 94-96.
4. Yoskovitch A, Tewfik TL, Brouillette RT, Schloss MD, Der Kaloustian VM. Acute Airway Obstruction in Hunter Syndrome. Internat J Ped Otorhinolaryngol 1998; 44: 273-78.
5. Menéndez C, Zaldívar C, González M. Errores Innatos del Metabolismo. Enfermedades Lisosomales. Rev Cub Pediat 2002; 74 (1): 68-76.
6. Desnick RJ. Enzyme Replacement and Enhancement Therapies for Lysosomal Diseases. J Inher Metab Dis 2004; 27: 385-410.
7. Sáenz H, Barrera LA. La Terapia de Reemplazo Enzimático en el Tratamiento de Enfermedades Genéticas. Univ Scient 2003; 8 (2): 31-42.
8. Landázuri P. Clonación Del cDNA de la Iduronato 2-Sulfato Sulfatasa Humana. Expresión de la Enzima en Escherichia coli y Pichia pastoris. Instituto de Errores Innatos del Metabolismo. Tesis Doctoral. 2002. Pontificia Universidad Javeriana. Bogotá, D.C. 115 p.
9. Chen Y, Jin M, Egborge T, Coppola G, Andre J, Calhoun DH. Expression and Characterization of Glycosylated and Catalytically Active Recombinant Human a-Galactosidase A Produced in Pichia pastoris. Prot Exp Purif 2000; 20: 472.84.
10. Files DA, Ogawa M, Scaman CH, Baldwin SA. Pichia pastoris Fermentation Process for Producing High-Levels of Recombinant Human Cystatin-C. Enz Microbiol Technol 2001; 26: 335 40.
11. Sinclair G, Choy F. Synonymous Codon Usage Bias and the Expression of Human Glucocerebrosidase in the Methylotrophic Yeast, Pichia pastoris. Prot Exp Purif 2002; 26: 96-105.
12. Xie J, Zhoub Q, Dub P, Ganb R, Ye Q. Use of Different Carbon Sources in Cultivation of Recombinant Pichia pastoris for Angiostatin Production. Enz Microb Technol 2005; 36: 210-16.
13. Poutou RA, Córdoba H, Quevedo BE, Landázuri P, Echeverri OY, Sáenz H, Vanegas A, Acero J, Gónzalez A, Herrera J, Algesira N, Caicedo L, Barrera LA. Expresión de Iduronato 2 sulfato Sulfatasa Humana Recombinante (IDShr) en Pichia pastoris. Univ Scient 2005; 10 (1): 73 94.
14. Cereghino GP, Cereghino J, Ilgen C, Cregg J. Production of Recombinant Proteins in Fermenter Cultures of the Yeast Pichia pastoris. Curr Opin Biotechnol 2002; 13: 329-32.
15. Cereghino JL, Cregg JM. Heterologous Protein Expression in the Methylotrophic Yeast Pichia pastoris. FEMS Microbiol Rev 2000; 24: 45-66.
16. Fischer R, Drossard J, Emans N, Commandeur U, Hellwig S. Towards Molecular Farming in the Future: Pichia pastoris- Based Production of Single-Chain Antibody Fragments. Biotechnol App Biochem 1999; 30: 117-20.
17. Romanos M. Advances in The Use of Pichia pasrtoris for High- Level Gene Expression. Curr Opin Biotechnol 1995; 6: 527-33.
18. Córdoba H, N. A, Poutou RA, Barrera LA. Pichia pastoris una Alternativa para la Producción de Glicoproteínas Humanas de Uso Terapéutico. Estrategias de Fermentación. Rev Col Biotecnol 2003; 5 (2): 73-84.
19. Hollenberg CP, Gellissen G. Production of Recombinant Proteins by Methylotrophic Yeast. Curr Opin Biotechnol 1997; 8: 554-60.
20. Caplan S, Green R, Rocco J, Kurjanlt J. Glycosylation and Structure of the Yeast MF Factor Precursor Is Important for Efficient Transport Through the Secretory Pathway. J Bacteriol 1991; 173 (2): 627-35.
21. Kurjan J, Herskowitz I. Structure of a Yeast Pheromone Gene (MF alpha): a Putative alpha factor Precursor Contains Four Tandem Copies of Mature alpha-factor. Cell 1982; 30: 933-43.
22. Egger M, Davey G, Altman D. Systematic Reviews in Health Care: Meta-analysis in Context. 2003; London: BMJ Books. 3-13, 23-29, 211-223.
23. Glasziou P, Irwig L, Bain C, Colditz G. Systematic Reviews in Health Care: A Practical Guide. 1 ed. 2001; Cambridge: Cambridge University Press. 4-14, 27- 31, 45-46.
24 . Rosenberg W, Donald A, Radcliffe J. Evidence Based Medici91 ne: An Approach to Clinical Problem-Solving, in Evidence Based Health Care Workbook: Understanding Research: for Individual Learning, A Donald, Editor. 1999, BMJ Publishing Group: London. p. 17-19, 37-38.
25. Daniel WW. Bioestadística. Base para el Análisis de las Ciencias de la Salud. 3a ed. 2000; Mexico: Ed. Limusa S.A. 245- 252,269-276.
26. Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller WJ, Lipman D. Gapped BLAST And PSI-BLAST: A New Generation of Protein Database Search Programs. Nuc Aci Res 1997; 25: 3389-3402.
27. Wilson PJ, Meaney CA, Hopwood JJ, Morris CP. Sequence of the Human Iduronate 2 Sulfatase (IDS) Gene. Genom 1993; 17: 773-75.
28. Koganesawa N, Aizawa T, Masaki K, Matsuura A, Nimori T, Bando H, Kawano K, Nitta K. Construction of an Expression System of Insect Lysozyme Lacking Thermal Stability: The Effect of Selection of Signal Sequence on Level of Expression in the Pichia pastoris Expression System. Prot Eng 2001; 14 (9): 705-10.
29. Oka C, Tanaka M, Muraki M, Harata K, Suzuki K, Jigami Y. Human Lysozyme Secretion Increased by Alpha-factor Prosequence in Pichia pastoris. Biosci Biotechnol Biochem 1999; 63 (11): 1977-83.
30. Reverter D, Ventura S, Villegas V, Vendrell JX, Avilés FX. Overexpression of Human Procarboxypeptidase A2 in Pichia pastoris and Detailed Characterization of Its Activation Pathway. The J Biol Chem 1998; 273 (6): 3535.41.
31. Roy N, Padmanabhan S, Smith M, Shi L, Navre M, Das G. Expression of Human Gelatinase B in Pichia pastoris. Prot Exp Purif 1999; 16: 324-30.
32. Sen Gupta C, Dighe R. Hyperexpression of Biologically Active Human Chorionic Gonadotropin Using the Methylotropic Yeast, Pichia pastoris. J Mol Endocrinol 1999; 22: 273-83.
33. Vuorela A, Myllyharju J, Nissi R, Pihlajaniemi T, Kivirikko KI. Assembly of Human prolyl 4 hydroxylase and Type III Collagen in the Yeast Pichia pastoris: Formation of a Stable Enzyme Tetramer Requires Coexpression with Collagen and Sssembly of a Stable Collagen Requires Coexpression with prolyl 4-hydroxylase. The EMBO J 1997; 16 (22): 6702.12.
34. Wendeler M, Hoernschemeyer J, John M, Werth N, Schoeniger M, Lemm T, Hartmann R, Kessler H, Sandhoff K. Expression of the GM2-Activator Protein in the Methylotrophic Yeast Pichia pastoris, Purification, Isotopic Labeling, and Biophysical Characterization. Prot Exp Puri f2004; 34: 147-57.
35. Kjeldsen T, Frost Pettersson A, Hach M. The Role of Leaders in Intracellular Transport and Secretion of the Insulin Precursor in the Yeast Saccharomyces cerevisiae. J Biotechnol 1999; 75: 195-208.
36. Murasugi A, Tohma-Aiba Y. Comparison of Three Signals for Secretory Expresión of Recombinant Human Midkine in Pichia pastoris. Biosci Biotechnol Biochem 2001; 65 (10): 2291 93.
37. Murasugi A, Tohma-Aiba Y. Production of Native Recombinant Human Midkine in the Yeast, Pichia pastoris. Prot Exp Purif 2003; 27: 244-52.
38. Kjeldsen T, Andersen A, Hach M, Diers I, Nikolajsen J, Markussen J. a-Factor Pro-Peptide N-linked Oligosaccharides Facilitate Secretion of the Insulin Precursor in Saccharomyces cerevisiae. Biotechnol App Biochem 1998; 27: 109-15.
39. Kjeldsen T, Pettersson AF, Hach M. Secretory Expression and Characterization of Insulin in Pichia pastoris. Biotechnol App Biochem 1999; 29: 79-86.
40. Liu SH, Chou W, Sheu C, Chang M. Improved Secretory Production of Glucoamylase in Pichia pastoris by Combination of Genetic Manipulations. Biochem Biophys Res Commun 2005; 326: 817-24.
41. Sreekrishna K, Brankamp R, Kropp K, Blankenship D, Tsay J-T, Smith P, Wierschke J, Subramaniam A, Birkenberger L. Strategies for optimal Synthesis and Secretion of Heterologous Proteins in the Methylotrophic Yeast Pichia pastoris. Gene 1997; 190: 55-62.
42. Cappai R, Mok SS, Galatis D, Tucker D, Henry A, Beyreuther K, Small D, Masters C. Recombinant Human Amyloid Precursor- Like Protein 2 (APLP2) Expressed in the Yeast Pichia pastoris Can Stimulate Neurite Outgrowth. FEBS Let 1999; 442: 95-98.
43. Pham C, Thomas D, Mercer J, Ley T. Production of Fully Active Recombinant Murine Granzyme B in Yeast. The J Biolo Chem 1998; 273 (16): 1629-33.
44. Goda S, Takano K, Yamagata Y, Katakura Y, Yutani K. Effect of NTerminal Residues on the Stability and Folding of Human Lysozyme Expressed in Pichia pastoris. Prot Eng 2000; 13 (4): 299 307.
45. Malmgren H, Calberg BM, Peterson U, Bondeson ML. Identificatión of an Alternative Transcript from the Human Iduronate 2- Sulfatase. Genomics 1995; 29: 291-93.
46. Invitrogen. Pichia Expression Kit. Protein Expression. A Manual of Methods for Expression of Recombinant Proteins in P. pastoris.Cat. No. K1710-01. 1996; California: Invitrogen.
47. Parodi A. Reglucosylation of Glycoproteins and Quality Control of Glycoprotein Folding in the Endoplasmic Reticulum of Yeast Cells. Biochim Biophys Acta 1999; 1426: 287-95.
48. Kowalski J, Parekh R, Mao J, Wittrup K. Protein Folding Stability Can Determine the Efficiency of Escape from Endoplasmic Reticulum Quality Control. The J Biol Chem 1998; 273 (31): 19453-58.
49. Dalbey RE, Lively MO, Bron S, Van Diijl JM. The Chemistry and Enzymology of the Type I Signal Peptidases. Prot Sci 1997; 6: 1129-38.
50. Peña O, Sosa A, Echeverri O, Sáenz H, Barrera LA. Producción de Anticuerpos Policlonales IgG Contra la Proteína Iduronato-2- sulfato Sulfatasa y Desarrollo de un Sistema de Detección para IDS Humana Recombinante. Bioméd 2005; 25: 181-88.