Diferenciación de especies de Rhodococcusmediante una prueba de PCR-RFLP basada en los genes codificantes para la subunidad 16S ribosomal
Paula Pavía1, Camila Calderon1, Concepción Puerta Ph.D.1
1Laboratorio de Parasitología Molecular, Departamento de Microbiología, Pontificia Universidad Javeriana.
Correspondencia: cpuerta@javeriana.edu.co
Recibido:11-08-05 / Aceptado:01-09-05
Palabras clave: Rhodococcus, R. equi, R. rhodnii, PCR-RFLP.
Key Words: Rhodococcus, R. equi, R. rhodnii, PCR-RFLP.
Por otra parte, dentro del género Rhodococcus también se encuentran bacterias simbiontes de insectos, tales como Rhodococcus rhodnii (5) y Rhodococcus triatomae (6), microorganismos que han sido encontrados en el intestino de los insectos triatominos Rhodnius prolixus y Triatoma spp., vectores transmisores de Trypanosoma cruzi, el agente causal de la enfermedad de Chagas.
Otros actinomicetos tales como Nocardia spp., Gordonia spp. y R. equi-.like., han sido encontrados en el intestino de los triatominos Triatoma infestans, Triatoma sordida y Panstrongylus megistus, respectivamente (7). Las bacterias simbiontes son importantes para estos insectos ya que les suplen nutrientes como aminoácidos, vitaminas y ácidos nucleicos, importantes en la nutrición de los insectos (8). Por lo tanto, su identificación y diferenciación constituye el primer paso en el uso potencial de las mismas como vehículos para el control de la enfermedad de Chagas (9,10).
Para la identificación de R. equi y otras especies del género, en general la clínica del paciente, su situación inmunológica, el origen de la muestra, el tiempo de crecimiento de dicha bacteria, la morfología de la colonia y las características fenotípicas de la cepa, pueden contribuir a realizar un diagnóstico presuntivo. En cuanto al diagnostico diferencial, la prueba más clara es la producción de factor Equi. Sin embargo, la diferenciación definitiva entre las especies de Rhodoccoccus y otros actinomicetos presentes en muestras clínicas, no se facilita por pruebas microbiológicas convencionales. Es así como se han estudiado otras pruebas de identificación y diferenciación como son las moleculares (3). Dentro de este contexto, en este trabajo se desarrolló una prueba de PCR, seguida de un ensayo de Polimorfismo de Longitud de Fragmentos de Restricción (PCR-RFLP), para diferenciar especies de Rhodococcus tomando como ejemplo las especies R. equi y R.rhodnii.
Bacterias: se estudiaron las siguientes especies bacterianas: R. rhodnii, aislado en el laboratorio de Parasitología Molecular (PUJB), a partir del triatomino vector R .prolixus y, R. equi (CECT 555), cepa amablemente cedida por la Colección Española de Cultivos Tipo de la Universidad de Valencia, España. Dichas bacterias fueron cultivadas en medio BHI, agar sangre y agar Corynebacterium a 37 y 26 °C. También se utilizaron otras bacterias como Escherichia coli, Acinetobacter baumani, Salmonella, Klebsiella, Lactobacillus, Citrobacter y Moraxella.
Extracción de ADN: para obtener el ADN de las bacterias se utilizó el método descrito por Shoemaker et al. (1986), con algunas modificaciones. Las bacterias fueron recolectadas por centrifugación a 10.000 rpm durante 15 minutos (min), a partir de un cultivo en medio BHI líquido. El botón bacteriano fue resuspendido en TE 1X (Tris-HCl 10 mM, pH 8,0, EDTA 1mM, pH 8,0) y lavado dos veces en la misma solución. Luego, el botón bacteriano fue resuspendido en 200 ml de TE 1X y tratado con 2 mg/ml de lisozima e incubado a 37 °C por 30 min. Posteriormente, se le adicionó SDS al 1% y 100 mg/ml de Proteinasa K e incubó a 65 °C por 4 horas (h). Tras adicionar 84 ml de NaCl 5 M y 60 ml de CTAB (bromuro de hexadecil trimetil amonio), la mezcla fue incubada a 65 °C durante 20 min. El ADN fue extraído con cloroformoalcohol isoamilico (24:1) volumen a volumen (v:v) y precipitado con 0,6 volúmenes de isopropanol. Finalmente, la madeja de ADN obtenida se lavó con etanol al 70%, se secó a temperatura ambiente (TA) y se resuspendió en TE 1X. La concentración del ADN obtenido se midió por espectrofotometría a 260 nm (12).
Condiciones de la reacción de PCR: el ADN bacteriano fue amplificado con los iniciadores: 16F (5.- GCTTAACACATGCAAG-3.) y 16R (5.-ACGGGCAGTGTGTACAAGACC-3.), correspondientes a las posiciones 45 a 61 y 1406 a 1386, respectivamente, del gen codificante para el ARN ribosomal 16S de E. coli (13). La PCR se realizó en un volumen final de 50 ml, según lo descrito por Hypsa y Dale, (1997) y el producto de amplificación fue visualizado mediante electroforesis horizontal en geles de agarosa al 1% coloreados con bromuro de etidio (12).
PCR-RFLP: los productos de amplificación obtenidos con cada una de las bacterias, fueron sometidos a digestión con diferentes endonucleasas según especificaciones de la casa comercial para cada enzima.
Los perfiles de restricción obtenidos fueron visualizados mediante electroforesis horizontal en geles de agarosa al 1% coloreados con bromuro de etidio (12).

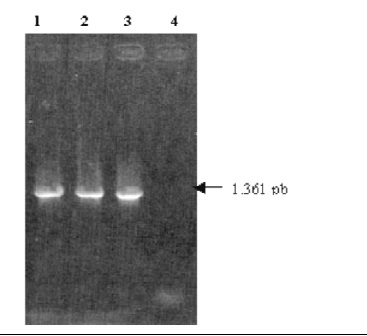
Figura 1. Resultados de la PCR 16SF/R No. 1. Electroforesis en gel de agarosa al 1% coloreado con bromuro de etidio conteniendo 10 ml del producto amplificado. R. equi (1), R. rhodnii (2), E. coli (3) y en el control negativo se usó agua destilada como templado (4).
Análisis in silico: se determinó mediante el programa TACG versión 3.2 (http:workbench.sdsc.edu) el patrón de restricción diferencial del gen 16S ARNr de R. equi, R. rhodnii y de 14 especies de Rhodococcus, 3 de Nocardia y 1 de Tsukamurella.
Ensayo de PCR 16S F/R
Una vez obtenido el ADN de R. equi, R. rhodnii y E. coli, este fue amplificado con los iniciadores conservados para los genes 16S ARNr, obteniendo el producto de amplificación esperado de 1361 pb, Figura
1. Este mismo ensayo de PCR, se realizó con un nú- mero mayor de bacterias, obteniendo de igual forma el producto de amplificación esperado, Figura 2.
Ensayos de PCR-RFLP
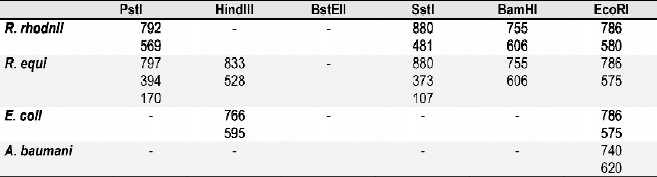
A partir de la digestión de los productos de amplificaci ón de las bacterias estudiadas con enzimas de restricción, se logró determinar el patrón de restricción diferencial entre R. equi, R. rhodnii y otros cocobacilos con las enzimas BamHI, HindIII, PstI, SstI, y EcoRI, Tablas 1 y 2 y Figuras 3-5.
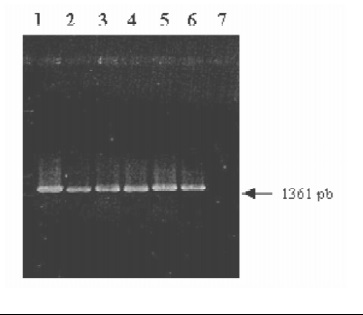
Figura 2. Resultados de la PCR 16SF/R No. 2. Electroforesis en gel de agarosa al 1% coloreado con bromuro de etidio conteniendo 10 ml del producto amplificado. Salmonella (1), Klebsiella (2), Acinetobacter (3), Lactobacillus (4), Citrobacter (5), Moraxella (6) y en el control negativo se usó agua destilada como templado (7).
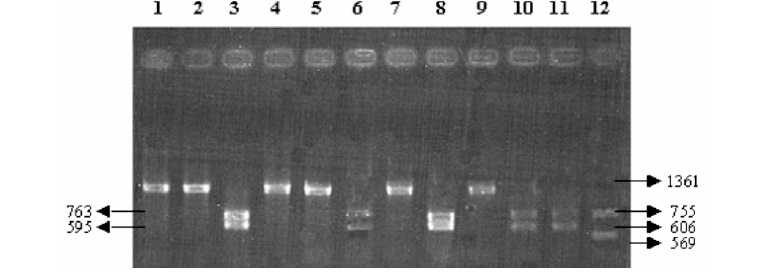
Figura 3. Resultados del perfil de restricción diferencial entre R. equi, R. rhodnii y E. coli. Electroforesis en gel de agarosa al 2% coloreado con bromuro de etidio conteniendo 15 ml del producto de digestión. E. coli sin digerir (1), perfil de restricción para E. coli: BamHI (2), HindIII (3) y PstI (4), R. rhodnii sin digerir (5), perfil de restricción para R. rhodnii: BamHI (6), HindIII (7) y PstI (8), R. equi sin digerir (9), perfil de restricción para R. equi: BamHI (10), HindIII (11) y PstI (12). Los tamaños de los fragmentos se indican en pb.
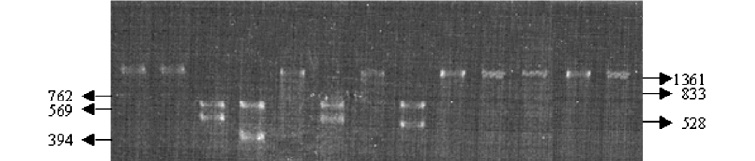
Figura 4. Resultados del perfil de restricción diferencial entre R. equi, R. rhodnii y otros cocobacilos. Electroforesis en gel de agarosa al 2% coloreado con bromuro de etidio conteniendo 15 ml del producto de digestión. E. coli sin digerir (1), perfil de restricción con la enzima PstI para: E. coli (2), R. rhodnii, (3), R. equi (4) y A. baumani (5), perfil de restricción con la enzima HindIII para: E. coli (6), R. rhodnii, (7), R. equi (8) y A. baumani (9), perfil de restricción con la enzima BstEII para: E. coli (10), R. rhodnii, (11), R. equi (12) y A. baumani (13). Los tamaños de los fragmentos se indican en pb.
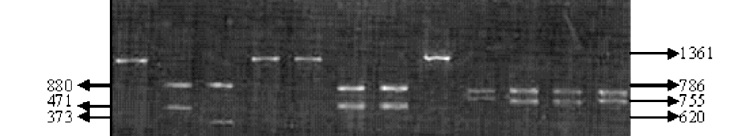 Figura 5. Resultados del perfil de restricción diferencial entre R. equi, R. rhodnii y otros cocobacilos. Electroforesis en gel de agarosa al 2% coloreado con bromuro de etidio conteniendo 15 ml del producto de digestión. Perfil de restricción con la enzima SstI para: E. coli (2), R. rhodnii, (3), R. equi (4) y A. baumani (5), perfil de restricción con la enzima BamHI para: E. coli (6), R. rhodnii, (7), R. equi (8) y A. baumani (9), perfil de restricción con la enzima EcoRI para: E. coli (10), R. rhodnii, (11), R. equi (12) y A. baumani (13). Los tamaños de los fragmentos se indican en pb.
Figura 5. Resultados del perfil de restricción diferencial entre R. equi, R. rhodnii y otros cocobacilos. Electroforesis en gel de agarosa al 2% coloreado con bromuro de etidio conteniendo 15 ml del producto de digestión. Perfil de restricción con la enzima SstI para: E. coli (2), R. rhodnii, (3), R. equi (4) y A. baumani (5), perfil de restricción con la enzima BamHI para: E. coli (6), R. rhodnii, (7), R. equi (8) y A. baumani (9), perfil de restricción con la enzima EcoRI para: E. coli (10), R. rhodnii, (11), R. equi (12) y A. baumani (13). Los tamaños de los fragmentos se indican en pb.
Tabla 1. Perfil de restricción diferencial entre R. equi, R. rhodnii y E.coli

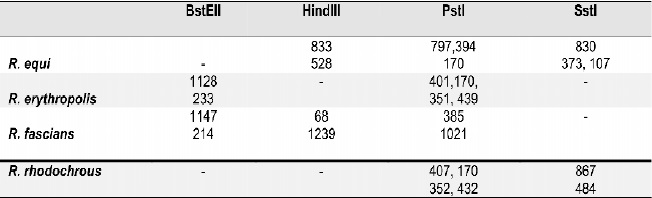
Tabla 2. Perfil de restricción diferencial entre R. equi, R. rhodnii y otros cocobacilos.
PCR-RFLP in silico
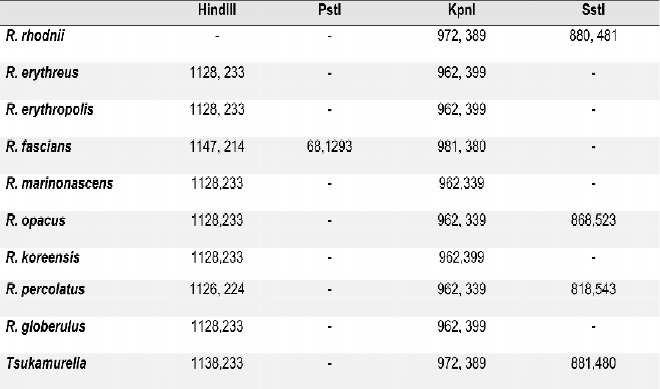
Se logró determinar por análisis in silico un algoritmo de diferenciación entre R. equi y R. rhodnii y entre estas dos especies y otras especies de Rhodococcus, Nocardia y Tsukamurella, Tablas 3-5. Los resultados obtenidos confirman los hallazgos exprimentales.
La PCR, seguida de un ensayo de Polimorfismos de Longitud de Fragmentos de Restricción (PCRRFLP) propuesta en este trabajo, es una prueba alternativa para la diferenciación entre distintas especies de Rhodococcus. Esta prueba combina la ventaja de la elevada conservación de la secuencia de nucleótidos de los genes ribosomales 16S entre distintos géneros bacterianos, con la especificidad de los polimorfismos de restricción, los cuales evidencian la variabilidad entre las secuencias amplificadas permitiendo la identificación a nivel de especie.
Con esta prueba se pudo determinar un perfil de restricción diferencial entre R. equi, R. rhodnii y otros cocobacilos. Debido al pleomorfismo y variabilidad que presentan las especies de Rhodococcus con ciertos colorantes, pueden ser confundidos con micobacterias de crecimiento rápido o con el género Acinetobacter (3). Es así como, en este trabajo se incluyó en el análisis otras especies bacterianas como Acinetobacter baumani y Escherichia coli, obteniéndose un patrón diferencial de estas especies con respecto a R. equi y R. rhodnii. El análisis de PCR-RFLP in silito permitió confirmar los anteriores resultados, así como también diferenciar estas bacterias de otras especies de Rhodococcus, Nocardia y Tsukamurella, las cuales también pueden ser aisladas a partir de diferentes muestras clínicas (2, 3).
Por otra parte, la PCR-RFLP, mostró ser una herramienta valiosa para la identificación y diferenciación de R. rhodnii, bacteria simbionte en el insecto triatomino R. prolixus, vector transmisor del parásito Trypanosoma cruzi, agente causal de la enfermedad de Chagas (5-10).
Tabla 3. Algoritmo de diferenciación de R. equi y R. rhodnii con otros Rhodococcus
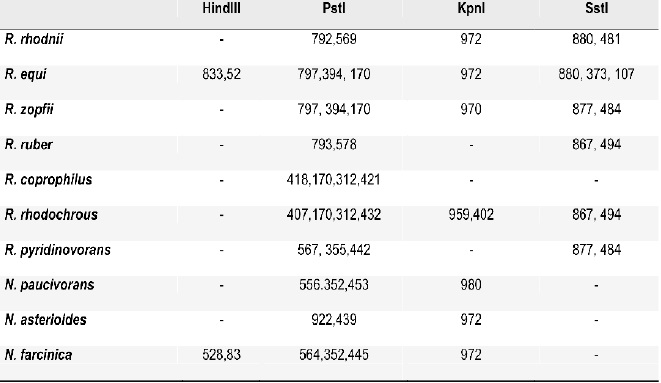
Tabla 4. Algoritmo de diferenciación de R. equi y R. rhodnii con otros Rhodococcus
Por lo tanto, esta prueba puede ser utilizada en la búsqueda, identificación y diferenciación de bacterias simbiontes en otras especies de triatominos.
Tabla 5. Algoritmo de diferenciación de R. equi y R. rhodnii con otros Rhodococcus
Adicionalmente, diferentes especies de Rhodococcus se han considerado de gran importancia porque son utilizadas a nivel de control ambiental y en la industria biotecnológica (14). Uno de los ejemplos es el uso de R. rhodochrous en la producción de acrilamida a nivel industrial (15). Teniendo en cuenta lo anterior, la prueba de PCR-RLFP propuesta en este trabajo puede resultar de gran ayuda en la identificación de las diferentes especies utilizadas a nivel industrial.
Finalmente, se recomienda aplicar este ensayo a partir de muestras biológicas y evaluar su aplicabilidad con las diferentes especies de Rhodococcus aisladas de muestras clínicas y ambientales.
A la Dra. Alba Alicia Trespalacios, directora del Cepario y del Laboratorio de Microbiología del Departamento de Microbiología de la Pontificia Universidad Javeriana, por su asesoría en la identificación bioquímica de los microorganismos estudiados y por facilitar la cepa ATCC de Rhodococcus equi.
1. Murria P, Rosenthal K, Kobayashi G. Nocardia, Rhodococcus and related Actinomycetes. En: Medical Microbiology, Tercera edición, Editorial Mosby, USA 1998.
2. Kedlaya I, Ing MB, Wong SS. Rhodococcus equi infections in immunocompetent hosts: case report and review. Clin Infect Dis 2001; 32: 39-46.
3. Weinstock DM, Brown AE. Rhodococcus equi: an emerging pathogen. Clin Infect Dis 2002; 34:1379-1385.
4. Prescott JF. Rhodococcus equi: an animal and human pathogen. Clin Microbiol Rev 1991; 4: 20 34.
5. Hill P, Campbell JA, Petrie LA. Rhodnius prolixus and its symbiotic actinomycete: a microbiological, physiological and behavioural study. Proc R Soc Lond B, Biol Sci Sp 1976; 194: 501-525.
6. Yassin AF. Rhodococcus triatomae sp. nov., isolated from a bloodsucking bug. Int J Syst Evol Microbiol 2005; 55: 1575-1579.
7. Eichler S, Schaub, GA. Development of symbionts in triatomine bugs and the effects of infections with trypanosomatids. Exp Parasitol 2002; 100: 17-27.
8. Beard C, Cordon-Rosales C, Durvasula R. Bacterial symbionts of the triatominae and their potential use in control of Chagas disease transmission. Annu Rev Entomol 2002; 47: 123-141.
9. Beard C, Mason P, Aksoy S, Tesh R, Richards F. Transformation of an insect symbiont and expression of a foreign gene in the Chagas´ disease vector Rhodnius prolixus. Am J Trop Med Hyg 1992; 46: 195 200.
10. Dotson EM, Plikaytis B, Shinnick TM, Durvasula RV, Beard CB. Transformation of Rhodococcus rhodnii, a symbiont of the Chagas disease vector Rhodnius prolixus, with integrative elements of the L1 mycobacteriophage. Infect Genet Evol 2003; 3: 103-109.
11. Shoemaker SA, Fisher JH, Jones WD Jr. Scoggin CH. Restriction fragment analysis of chromosomal DNA defines different strains of Mycobacterium tuberculosis. Am Rev Respir Dis 1986; 134: 210-213.
12. Sambrook JF, Russell DW. Molecular Cloning, A Laboratory Manual. Tercera edición, Cold Spring Harbor Laboratory Press, New York 2001.
13. Hypsa V, Dale C. In vitro culture and phylogenetic analysis of .Candidatus Arsenophonus triatominarum. an intracellular bacterium from the triatomine bug, Triatoma infestans. Int J Syst Bacteriol 1997; 47: 1140-1144.
14. Bell KS, Philp JC, Aw DW. The genus Rhodococcus. J Appl Microbiol 1998; 85: 195-210.
15. Yamada H, Kobayashi M. Nitrile hydratase and its application to industrial production of acrylamide. Biosci Biotechnol Biochem 1996; 60: 1391-1400.