Influencia de la fuente de carbono sobre la expresión de proteínas AOX1-reguladas en Pichia pastoris
Raúl A. Poutou BQ, MSc.1,2, Balkys E. Quevedo Ing.Q, MSc.2, Henrry A. Córdoba IngQ., MSc.1,3, Homero Sáenz Biol MSc.1,4, Luis A. Barrera BQ, PhD.1
1Instituto de Errores Innatos del Metabolismo
2Grupo de Biotecnología Ambiental e Industrial, Departamento de Microbiología
3Departamento de Química, Pontificia Universidad Javeriana, Bogotá D.C., Colombia.
4Decanato de Medicina, Universidad Centroccidental Lisandro Alvarado, Barquisimeto, Venezuela.
Correspondencia: E-mail: abarrera@javeriana.edu.co
Recibido: 13-05-2005 / Aceptado:27-05-2005
La levadura Pichia pastoris constituye un excelente modelo para la expresión de proteínas heterólogas, lo que se refleja en el gran número de proteínas que han sido obtenidas en este modelo biológico, bajo el control del promotor AOX1. Esto implica que el número de peroxisomas y la enzima alcohol oxidasa es regulada como respuesta a la inducción por metabolitos como el metanol, el glicerol, el etanol, el acetato y vías metabólica como la b-oxidación. En esta revisión, se discuten aspectos relacionados con el metabolismo de estos compuestos y su posible influencia en la producción de proteínas recombinantes, específicamente la Iduronato 2- sulfato sulfatasa humana (IDSh).
Palabras clave: Pichia pastoris, AOX, represión catabólica, autofagia, fuente de carbono, peroxisoma
Influence of carbon source over the expression of AOX1-regulated proteins in Pichia pastoris: The Pichia pastoris yeast is an excellent model for the expression of heterologous protein, thus could be deduce from the greate number of proteins that has been obtain in this biological model under the control of AOX1 promoter. This means that peroxisomes number and the enzyme alcohol oxidase are regulate as response to the induction of different metabolites such as methanol, glycerol, ethanol, acetate and metabolic pathways like the b- oxidation. In this review, aspects related with the metabolism of this compounds and its possible influence in the production of recombinant proteins are discuss, specifically the human Iduronate 2-sulfate sulfatase (hIDS).
Key words: Pichia pastoris, AOX, catabolic repression, autophagy, carbon source, peroxisome.
Las levaduras han sido una de las células hospedero más comúnmente empleadas para la producción de proteínas heterólogas.Al comienzo, la aplicación se centró en el uso de las especies mejor caracterizadas como Saccharomyces cerevisiae, con el objetivo de producir proteínas recombinantes. Sin embargo, en antilde;os recientes levaduras diferentes a S. cereviciae se han hecho más accesibles gracias a las estrategias modernas de genética molecular y por lo tanto, se han derivado aplicaciones considerables en el campo de la biotecnología. En este sentido, los modelos de expresión de genes heterólogos en Hansenula polymorpha y Pichia pastoris (levaduras no convencionales) han sido utilizados para producir altos niveles de proteínas recombinantes, tanto intra como extracelulares, con ventajas significativas sobre las levaduras convencionales y las líneas celulares de mamíferos e insectos (1-3).Wolf (4) sentilde;ala que, el término no convencional hace alusión a todos los géneros de levaduras excepto Saccharomyces y Schizosaccharomyces. Sin embargo, es importante considerar que con el paso del tiempo y la profundización en el conocimiento y manipulación de las levaduras, que hoy se consideran no convencionales, estas podrían llegar a ser reconocidas como las primeras (4).
Una de las categorías incluidas dentro de las levaduras no convencionales son las levaduras metilotróficas, entre la que encontramos a Pichia pastoris, Pichia angusta (Hansenula polymorpha), Pichia methanolica (Pichia pinus) y Candida boidini entre otras. El término metilotrófica hace referencia a las cepas capaces de emplear el metanol como única fuente de carbono y de energía; adaptación relacionada con la presencia de genes que codifican para enzimas involucradas en la degradación del mismo (5).
Varios estudios han demostrado que el metabolismo del metanol en levaduras metilotróficas, requiere de la expresión de un grupo de enzimas, entre las cuales la alcohol oxidasa (AOX I) (E.C.1.1.3.13) se hace indetectable en células cultivadas sobre fuentes de carbono tales como glucosa; sin embargo, esta enzima puede llegar a ser más del 30% del total de las proteínas solubles en células crecidas en metanol.
Estos estudios permitieron anticipar que la síntesis deAOX I podría ser regulada a nivel transcripcional y que el promotor para el gen podría ser útil para controlar la expresión de genes foráneos. Así, bajo el control del promotorAOX, los genes foráneos pueden ser mantenidos en un modo apagadoencendido, apagado en fuentes de carbono diferentes al metanol y encendido en un medio con metanol (6, 7).
Algunas de las especies de Pichia son consideradas de importancia biotecnológica, por ejemplo P. stipitis (anamorfa Candida shehatae) se ha destacado en la fermentación de xilosa (componente abundante en la biomasa vegetal); además, P. pastoris y P. angusta han sido empleadas en la expresión de proteínas recombinantes (4, 8); de otro lado P. nakazawae es productora de amilasa, mientras que P. guilliermondi produce riboflavina y algunas mutantes de P. methanolica deficientes de la enzima acetil-CoA sintetasa (E.C. 6.2.1.1) han sido empleadas como biosensores (5, 9-12).
Las levaduras metilotróficas fueron aisladas por primera vez en 1969 y su atractivo principal fue la producción de proteína celular simple (SCP) (13-15).
El género Pichia pertenece a la familia Saccharomycetaceae, órden Saccharomycetales, clase Saccharomycetes, filo Ascomycota, reino Fungi. Esta levadura presenta reproducción sexual con producción de ascosporas, en células productoras de esporas (4 esporas por asca con baja viabilidad) que originan dos formas, un zigoto o una forma patogénica de una célula somática simple. En medio sólido forma colonias no filamentosas de color blanco o color crema. Al microscopio se pueden observar múltiples yemas.
El género Pichia posee muchas especies, asociadas con insectos, que han sido aisladas de lodos fundidos y galerías de coleópteros. Algunas especies cactofílicas, son habitantes específicos de cactus. De otro lado se han encontrado formado asociaciones con drosófila; estas asociaciones con insectos presentan ascosporas en forma de sombrero o hemiesférica (saturno-esporas), morfología común para un amplio número de hongos no relacionados entre sí. Especies de este género pueden ser encontradas en diversos ambientes, incluyendo salmuera y fluidos de curtiembres. Las especies que presentan Saturno-esporas han sido reasignadas a los géneros Williopsis y Saturnispora, y aquellas especies con esporas esféricas planas pasaron al género Debaryomyces.
Muy cercano al género Yamadazyma el género Hyphopichia es considerado un sinónimo del género Pichia (5). Dentro del género Pichia se encuentran especies tanto fermentativas como no fermentativas y se han encontrado varios tipos de coenzimas Q (CoQ7, CoQ8 y CoQ9) como intermediarias en la cadena de trasporte electrónico, lo que muestra que el género es muy heterogéneo; sin embargo, algunas de sus especies han sido transferidas de forma prematura al género Yamadazyma. Hasta 1996 este género había sido enriquecido con la transferencia de algunas especies del género Hansenula.
Los medios y los protocolos para el crecimiento de P. pastoris fueron desarrollados en la década de los 70´s por la Philips Petroleum Company (4, 16), como herramienta biológica para utilizar el metanol, (fuente de carbono de bajo costo), en la obtención de proteínas de alta calidad para la alimentación del ganado (17). Varios sistemas de cultivo fueron formulados para esta levadura, los cuales generaron concentraciones de biomasa seca alrededor de 130g/l y productividades de 10g l-1 h-1 (18). Desafortunadamente la libra de SCP resultó ser mucho más costosa que la libra de soya. Por tal motivo, la compantilde;ía dirigió sus esfuerzos al desarrollo de sistemas de expresión de proteínas recombinantes (19, 20); sistemas que en la actualidad han servido para la expresión de una gran cantidad de proteínas foráneas (1, 8, 20-24).
El sistema de producción de proteínas recombinantes, desarrollado por la Philips Petroleum Company resultó ser más eficiente y económico, comparado con los sistemas bacterianos y el cultivo de células de mamíferos e insectos. Este sistema posee varias características que se suman a las altas densidades de crecimiento y a la productividad referida con anterioridad; estas características son: (i) el costo de los medios, equipo e infraestructura de los cultivos de levaduras son mucho más económicos que los de las células de mamíferos e insectos, (ii) el sistema de expresión de proteínas disentilde;ado, es inducido por adición de metanol y generalmente es integrado al cromosoma de la levadura para ganar estabilidad en la construcción genética, (iii) este microorganismo realiza modificaciones postraduccionales como la glicosilación, adicionando un patrón de azúcares de tamantilde;o pequentilde;o y muy similar al humano, (iv) la posibilidad de secretar de forma directa la proteína clonada, lo que significa en términos prácticos un paso de pre-purificación y (v) esta levadura no produce endotoxinas por lo que podría considerarse un microorganismo seguro (GRAS) (2, 8, 24-26).
Los peroxisomas son organelos que se encuentran presentes en todas las células eucariotas y se caracterizan por la presencia de enzimas como la catalasa (E.C. 1.11.1.7) y al menos una oxidasa generadora de peróxido de hidrógeno (H2O2). Estos organelos están constituidos por una membrana simple y pueden variar considerablemente de tamantilde;o, cantidad y contenido enzimático, dependiendo del organismo, el tipo de célula y las condiciones ambientales. Teniendo en cuenta que los peroxisomas no poseen ácidos nucleicos ni ribosomas, todas las proteínas del peroxisoma están codificadas en el ADN nuclear y deben ser sintetizadas e importadas postraduccionalmente a los peroxisomas, gracias a las secuencias sentilde;ales para la importación .peroxisomal targeting signals. (PTS). Hasta 1999 dos clases de PTSs se habían caracterizado. La primera y más común la PTS1, es una sentilde;al compuesta por los tres primeros aminoácidos de la región C-t de muchas de las proteínas peroxisomales de animales, plantas y levaduras (SKL); la segunda sentilde;al (PTS2) conformada por la secuencia (RLX5H/QL), secuencia que se localiza cerca de la región N-t de las proteínas peroxisomales en mamíferos y levaduras. En levaduras la biogénesis de los peroxisomas (proliferación) es inducida marcadamente por la utilización de ciertas fuentes de carbono tales como el metanol y el ácido oléico (27-31).
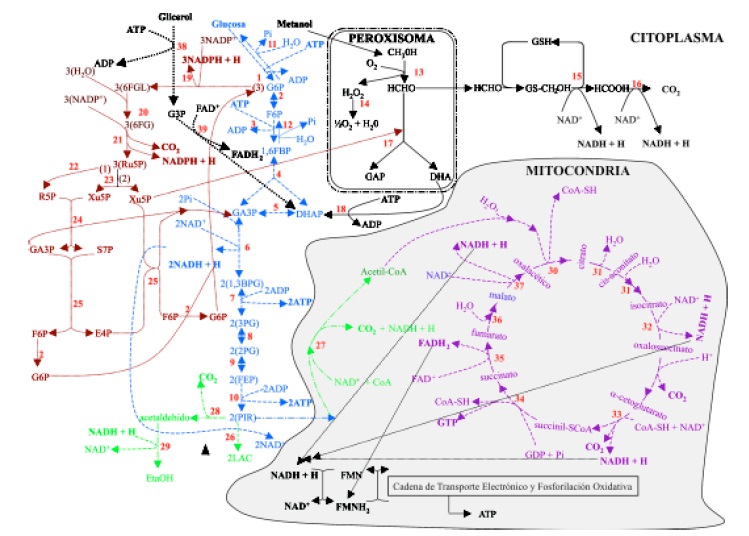
El metabolismo del metanol se inicia en el interior de los peroxisomas. El metabolismo comienza cuando el metanol es oxidado, en presencia de oxígeno, por la enzima alcohol oxidasa, produciendo formaldehído y H2O2. El peróxido de hidrógeno es tóxico para la célula, por tanto es retenido en el peroxisoma hasta que la catalasa lo convierte en H2O y O2. Una parte del formaldehído es convertido en dihidroxiacetona (DHA) y gliceraldehido fosfato (GAP) a través de la acción de la transcetolasa dihidroxiacetona sintasa (E.C.2.2.1.3). La DHA es liberada al citosol y fosforilada por la dihidroxiacetona quinasa (E.C. 2.7.1.29) para formar DHAP; esta molécula puede experimentar dos reacciones, la reacción de interconversión de triosas fosfatos para formar GAP gracias a la acción de la enzima triosa fosfato isomerasa (E.C. 5.3.1.1) y alimentar así la segunda fase de la vía glucolítica; la otra reacción y al parecer la más descrita por los autores es la condensación con una molécula de GAP para formar fructosa 1,6-bifosfato (1,6FBP) y luego fructosa 6-fosfato (F6P), en estas transformaciones intervienen las enzimas fructosa 1,6-bifosfato aldolasa (E.C.4.1.2.13) y fructosa 1,6-bifosfatasa (E.C.3.1.3.11) respectivamente; esta última hace parte del proceso conocido como gluconeogénesis (13, 24, 32).
De forma alternativa, la fructosa 6-fosfato (F6P) puede formar biomasa o incorporarse a la vía de las pentosas fosfatos (vía de las pentosas) para formar xilulosa 5-fosfato (Xu5P) y realimentar el ciclo en DHAP; de igual manera la DHAP es regenerada varias veces lo que favorece la formación de biomasa.
El formaldehído restante de la oxidación del metanol difunde al citosol donde reacciona con glutatión a través de las enzimas formaldehído deshidrogenasa (E.C.1.2.1.1) y formato deshidrogenasa (E.C.1.2.1.2), para formar CO2 con desprendimiento de NADH+H (fuente de energía) (13, 24, 33) Figura 1.
Una vez que el glicerol entra a la célula es fosforilado por la glicerol quinasa (E.C.2.7.1.30) para formar glicerol 3-fosfato (G3P), posteriormente ocurre la oxidación del G3P a DHAP debido a la acción de la enzima FAD-dependiente glicerol-3-fosfato deshidrogenasa (E.C.1.1.99.5). Finalmente la DHAP continúa la segunda fase de la vía glicolítica y se obtiene el ácido pirúvico. Posteriormente el piruvato es oxidado a acetil-CoA y alimenta el ciclo de los ácidos tricarboxilicos (TCA) de donde se obtienen GTP, NADH + H y FADH2, estos dos últimos serán utilizados posteriormente para generar ATP a través de la cadena de transporte electrónico y fosforilación oxidativa (Figura 1) (33-36).
A partir del piruvato puede dispararse la fermentación alcohólica, lo que depende de la capacidad respiratoria y el ritmo metabólico de la glucólisis. En este proceso el piruvato es transformado a acetaldehído por la piruvato descarboxilasa (E.C.4.1.1.1) y éste es reducido para formar etanol, reacción catalizada por la enzima alcohol deshidrogenasa (E.C.1.1.1.1) (33, 37). Adicionalmente, el etanol puede ser empleado como fuente de carbono y energía, si se restablece la capacidad respiratoria de la levadura. En este caso el etanol sería transformado hasta acetaldehído por la alcohol deshidrogenasa y luego a acetato por la acetaldehído deshidrogenasa (E.C. 1.2.1.10) para que finalmente la acetil-CoA sintasa lo convierta en acetil-CoA (33).
Para la producción de proteínas heterólogas en P. pastoris, normalmente se emplea el esquema de tres fases en cultivos de alta densidad.

Figura 1. Mapa metabólico parcial de Pichia pastoris disentilde;ado por los autores para este artículo. números en color rojo: enzimas que participan en cada vía. (color azul): glucólisis y gluconeogénesis en el citoplasma, 1: hexoquinasa (E.C.2.7.1.1), 2: glucosa fosfato isomerasa (E.C.5.3.1.9), 3: 6-fosfofructo quinasa (E.C.2.7.1.11), 4: fructosa 1,6-bifosfato aldosa (E.C.4.1.2.13), 5: triosa fosfato isomerasa (E.C.5.3.1.1), 6: gliceraldehido fosfato deshidrogenasa (E.C.1.2.1.12), 7: fosfoglicerato quinasa (E.C.2.7.2.3), 8: fosfoglicero mutasa (E.C.2.7.5.3), 9: enolasa (E.C.4.2.1.11), 10: piruvato quinasa (E.C.2.7.1.40), 11: glucose 6-phosphatase (E.C.3.1.3.9), 12: fructosa 1,6- bifosfatasa (E.C.3.1.3.11). (color negro): degradación de metanol en el peroxisoma, 13: alcohol oxidasa (E.C.1.1.3.13), 14: catalasa (E.C. 1.11.1.7), 15: formaldehído deshidrogenasa (E.C.1.2.1.1), 16: formato deshidrogenasa (E.C.1.2.1.2), 17: dihidroxiacetona sintasa (E.C.2.2.1.3), 18: dihidroxiacetona quinasa (E.C. 2.7.1.29). (color marrón): vía de las pentosas fosfato, 19: glucosa 6-fosfato deshidrogenasa (E.C.1.1.1.49), 20: lactona hidrolasa (E.C.3.1.1.45), 21: 6-fosfogluconato deshidrogenasa (E.C.1.1.1.43), 22: ribosa fosfato isomerasa (E.C.5.3.1.6), 23: ribulosa fosfato 3-epimerasa (E.C.5.1.3.1 ), 24: transcetolasa (E.C.2.2.1.1), 25: transaldolasa (E.C. 2.2.1.2). (verde): reacciones no relacionmadas directamente con ninguna vía metabólica, 26: lactato deshidrogenasa (E.C.1.1.1.27), 27:
complejo de la piruvato deshidrogenasa (piruvato deshidrogenasa, dihidrolipoil-transcetilasa y dihidrolipoil-deshidrogenasa), 28: piruvato descarboxilasa (E.C.4.1.1.1), 29: alcohol deshidrogenasa (E.C.1.1.1.1). (morado): ciclo de krebs, 30: citrato sintasa (E.C. 4.1.3.7), 31: aconitasa (E.C.4.2.1.3), 32: isocitrato deshidrogenasa (E.C.1.1.1.41), 33: a-cetoglutarato deshidrogenasa (E.C.1.2.4.2), 34: succinil-CoA sintetasa (E.C.6.2.1.4), 35: succinato deshidrogenasa (E.C.1.3.99.1), 36: fumarasa (E.C. 4.2.1.2), 37: malato deshidrogenasa (E.C. 1.1.1.37). (color negro al interior de la mitocondria): cadena de transporte elctrónico. (color negro discontinua de punto grueso): Metabolismo del glicerol, 38: glicerol quinasa (E.C.2.7.1.30), 39: glicerol 3-fosfato deshidrogenasa (E.C.1.1.99.5).
En la primera fase la levadura recombinante es cultivada en medios salinos suplementados con un azúcar no fermentable como el glicerol (cultivo discontinuo). Una vez que el glicerol es consumido se inicia la fase de transición; en esta fase se alimenta glicerol en cantidades que mantengan el crecimiento exponencial limitado por sustrato. El consumo de productos secundarios como el acetato y el etanol generados durante el cultivo discontinuo son de gran importancia para preparar las células para la tercera fase o fase de inducción (37).
En la tercera fase generalmente se adiciona metanol en concentraciones limitadas, lo que debe resultar en la expresión de la proteína recombinante. Como en la mayoría de los trabajos realizados, la utilización de glicerol en la fase de crecimiento resulta en la producción de etanol y acetato (Figura 1). Inan y Meagher (37, 38), estudiaron el comportamiento en el crecimiento de P. pastoris al adicionar etanol y acetato durante la fase de inducción. En este estudio se demostró que P. pastoris es capaz de crecer en glicerol, metanol y etanol como únicas fuentes de carbono y energía; además, se demostró el crecimiento diáuxico cuando dos o tres de estas fuentes de carbonos se encontraban presentes en forma simultánea, especialmente en las mezclas etanol-metanol y etanolglicerol. De esta manera se pudo establecer la prioridad de utilización de las diferentes fuentes de carbono, glicerol > etanol > metanol (37).
Durante el crecimiento en la mezcla glicerol-etanol el consumo del glicerol precede la utilización del etanol, lo que genera la acumulación transiente de acetato una vez que se ha agotado el glicerol. La diauxia se genera durante la adaptación de la P. pastoris para crecer a expensas del acetato como única fuente de carbono (37) (Figura 1). Otro patrón de crecimiento diáuxico ha sido observado al crecer la levadura en la mezcla etanolmetanol, en este caso el metanol no es consumido hasta que se agota el etanol; sin embargo, la producción de acetato es 10 veces menor (37). En el caso de la mezcla glicerol-metanol no fue posible observar un comportamiento diáuxico evidente, a pesar de que el glicerol es consumido más rápidamente. En esta mezcla el consumo de metanol comienza antes que el consumo de glicerol termine, lo que indica que ciertos niveles de glicerol no reprimen (37) la expresión de la alcohol oxidasa. Cuando se emplea la mezcla de las tres fuentes de carbono la prioridad de utilización es la siguiente, glicerol > etanol > acetato (acumulado por la utilización del etanol) > metanol.
El glicerol es una fuente de carbono no fermentable y P. pastoris no es considerada una levadura fermentativa; sin embargo, el etanol se acumula durante el cultivo cuando se emplea una tasa alta de alimentación de glicerol. Este fenómeno ha sido reportado en varias ocasiones pero el mecanismo aún no ha sido explicado (39-41). Algunos autores han considerado que el etanol es metabolizado a acetaldehído y luego a acetato, el cual es asimilado como acetil-CoA; proceso que ha sido descrito para H. polymorpha y para P. methanolica (37, 42, 43).
La regulación del metabolismo del metanol en los organismos metilotróficos es un proceso complejo que incluye la síntesis, la activación y la degradación de las enzima involucradas. Estas enzimas son inducidas por metanol, formaldehído y formato (efectores positivos). Sin embargo, la actividad alcohol oxidasa es disminuida por la glucosa y el etanol (efectores negativos) a través de dos mecanismos de regulación, la represión y la inactivación por catabolito. El primero de estos mecanismos involucra el control sobre la síntesis deRNA y el segundo la inactivación o degración de enzimas. La formación de acetato como consecuencia de la utilización del etanol reprime la expresión de la alcohol oxidasa mientras que se agota (37).
En sentido general la represión por catabolito está mediada por la concentración intracelular de monofosfato cíclico de adenosina (AMPc), concentración que es inversamente proporcional a la concentración de glucosa en el medio de cultivo. Cuando la célula crece en presencia de glucosa el nivel intracelular de AMPc es bajo, a causa de que la glucosa inhibe la actividad adenilciclasa (E.C.4.6.1.1). Cuando la glucosa se consume totalmente aumenta la concentración de AMPc rápidamente y forma un complejo con la .proteína receptora de AMPc., complejo que actúa sobre el promotor AOX1, induciendo la utilización de otras fuentes de carbono (44).
El crecimiento de las levaduras metilotróficas en presencia de metanol como única fuente de carbono y energía trae como consecuencia la biogénesis de los peroxisomas. Cuando células crecidas en metanol son transferidas a medios de cultivos que contienen fuentes de carbonos diferentes como glucosa o etanol, los peroxisomas desaparecen rápidamente como consecuencia de una degradación activa, que involucra la degradación proteolítica de los peroxisomas (45-48).
Estudios realizados por Murray et al., (45), demostraron que la desaparición de los peroxisomas en P. pastoris después de 24h de bioconversión de etanol a acetaldehído confirma la inactivación por catabolito de la alcohol oxidasa; de la misma manera confirmaron, que la pérdida de los peroxisomas en células incubadas durante 48h en tampón fosfato en presencia de acetaldehído y O2, es causada por el acetaldehído. Se cree que la presencia del acetaldehído genera la necesidad de un metabolismo aeróbico. El papel del O2 hasta 1990, no era muy claro; sin embargo, las conclusiones de Murray (45) estaban basadas en observaciones realizadas en H. polymorpha, microorganismo en que se demostró la posibilidad prevenir la degradación de los peroxisomas cuando se inhibe el metabolismo energético a través de la incubación anaeróbica, por la adición de iones que inhiben la cadena respiratoria o cuando se transfieren las células a medios de cultivos con fuentes de carbono no metabolizables, como la deoxiglucosa (49).
También se sugiere que la utilización de células de P. pastoris para la bioconversión de etanol a acetaldehído mimetiza la situación en la que células crecidas en metanol son transferidas a un medio con etanol; bajo estas condiciones los peroxisomas no son requeridos, lo que dispara el inicio de una serie de eventos regulatorios relacionados con la degradación de los peroxisomas y con la proteólisis (autofagia) para generar nuevamente una mezcla de aminoácidos. Un aumento en la concentración de AMPc ocurre cuando aparecen las primeras moléculas de acetaldehído en presencia de O2, lo que activa las proteínas quinasas dependientes de AMPc. Estas proteínas fosforilan las enzimas del peroxisoma, lo que funciona como una marca o sentilde;al para la degradación proteolítica posterior (45).
En P. pastoris la inactivación por catabolito se completa alrededor de 21h después de que aparece la primera molécula libre de acetaldehído como resultado de la bioconversión de etanol (45).
Las células de mamíferos y las levaduras son capaces de responder a los cambios ambientales y al estrés nutricional con la activación de la degradación de proteínas a través de dos procesos conocidos como microautofagia y macroautofagia (49-51).
La microautofagia es el proceso donde los componentes celulares son rodeados por invaginación de la membrana lisosomal o por una protuberancia del lisosoma similar a un dedo o por una vacuola. El resultado es la formación de vesículas intralisosomales que contienen secuestrados los componentes celulares fosforilados y que serán degradados por las enzimas hidrolíticas (52).
La macroautofagia es el proceso donde los componentes celulares son secuestrados primeramente dentro de un autofagosoma originado por la invaginación del retículo endoplasmático rugoso. Entonces el autofagosoma se fusiona con un lisosoma o una vacuola liberando su contenido para que las hidrolasas ácidas actúen sobre los componentes marcados (fosforilados). Las enzimas citosólicas y peroxisomales involucradas en el metabolismo del metanol son sintetizadas cuando P. pastoris crece en presencia de metanol. Cuando se transfieren células adaptadas a crecer en metanol a un medio con glucosa o etanol, estas enzimas son secuestradas selectiva y rápidamente y degradadas dentro de las vacuolas de la levadura (52).
La alcohol oxidasa funcional, es un homooctámero, en el cual cada subunidad (~74kDa) contiene un enlace no covalente con nucleótidos de flavina (FAD+) como grupo prostético (7, 53, 54). Esta enzima codificada por el gen AOX1, presenta baja afinidad por el O2; afinidad que P. pastoris compensa con la producción de grandes cantidades de la misma (55). Estudios recientes mostraron la existencia de un segundo gen funcional de alcohol oxidasa en P. pastoris, (AOXII), el cual tiene 90% de homología en la región codificante con AOX1. Sin embargo esta enzima AOX II es sólo una pequentilde;a parte de la AOX producida por esta levadura (55-57).
La regulación de la expresión génica de AOX al parecer es mediada por represor, de forma tal que, cuando la levadura crece en presencia de glucosa o etanol no se sintetiza la enzima (represión por catabolito). La expresión de la enzima se induce cuando se crece en metanol, llegando a significar hasta el 30% de la proteína total producida por la célula (21, 55, 56). Esta regulación implica dos pasos, un mecanismo represión/derepresión o apagado/encendido (.on-off.), sumado a un mecanismo de inducción. La derepresión del AOX1 (ausencia de una fuente de carbono como glucosa, etanol o acetato) no es suficiente para que se exprese la enzima, aún en niveles basales. Entonces se requiere del agente inductor (metanol) para obtener niveles detectables de AOX (58). La pérdida del AOX1 significa por lo tanto la pérdida de la mayor parte de la actividad alcohol oxidasa (~80%), y resulta en el fenotipo conocido como Muts (utilización lenta del metanol y crecimiento pobre) (16). El fenotipo Mut+, es el fenotipo nativo y le permite a las células crecer en medio con metanol (1-3, 16, 58).
Estudios realizados en H. polymorpha han demostrado la presencia de monómeros inactivos de AOX en el citosol y que estos son exportados al peroxisoma (dependiendo de las PTS) donde la proteína es activada (formación del octámero y unión de FAD+) (59). Experimentos realizados .in vivo. e .in vitro. Demuestran que el ensamblaje del octámero activo no es un proceso espontáneo y que se requiere de ciertos factores de ensamblaje (proteínas auxiliadoras específicas) entre las que se encuentran factores de unión, factores de ensamblaje y factores de traslocación (60, 61); adicionalmente estos experimentos sugieren, que no es necesario un ambiente ácido como el de la matriz peroxisomal para el ensamblado de la AOX (59, 62).
Ozimek et al., (59), identificaron la proteína piruvato carboxilasa (E.C. 6.4.1.1) como la primera proteína que tiene una función esencial en el ensamblaje de la AOX en levaduras metilotróficas. La piruvato carboxilasa es una enzima anaplerótica que alimenta el ciclo de los ácidos tricarboxílicos (TCA) con oxalacetato obtenido a partir de piruvato. Las levaduras deficientes de esta enzima no pueden crecer en médios mínimos con glucosa, a menos que sean suplementados con aspartato o glutamato y en forma inesperada se demostró que las cepas de H. polymorpha y P. pastoris deficientes de esta enzima, son incapaces de crecer en metanol, independientemente de la presencia de aspartato o glutamato y que esta deficiencia en el crecimiento se debe a un bloqueo severo en el proceso de ensamblaje de la AOX. Los resultados de Ozimek et al. (59), fueron convincentes en que la proteína piruvato carboxilasa (no, la actividad enzimática) es necesaria para el ensamblaje de la alcohol oxidasa. Otros estudios (53, 59) .in vitro., han revelado la afinidad de la proteína piruvato carboxilasa y su interacción física con AOX (53, 59), sugiriendo que en levaduras metilotróficas la primera de estas proteínas juega un doble papel; de un lado la conocida y bien caracterizada función como enzima anaplerótica y del otro, un papel específico en el proceso de organización y ensamblaje de la alcohol oxidasa, posiblemente mediando la unión de FAD+ a los monómeros de AOX (53, 59).
Algunos trabajos han permitido proponer un sistema de expresión para la producción de proteínas recombinantes en P. pastoris (2, 8, 18, 58, 63), lo que ha generado el desarrollo de nuevos vectores de expresión, basado en que P. pastoris no presenta plásmidos nativos. Estos vectores se integran por recombinación homóloga al genoma de la levadura portando genes de complementación autotrófica como HIS4 que codifica para la enzima histidinol deshidrogenasa (E.C. 1.1.1.23), y son utilizados como marcadores de selección; se producen mutantes de crecimiento lento o rápido (Muts y Mut+) dependiendo del sitio de integración (AOX 1 o HIS respectivamente) (23, 64, 65).
Sibirny (66), estudió la inactivación de la alcohol oxidasa y la catalasa como consecuencia de la presencia de etanol y acetato en mutantes de P. methanolica, donde encontró que la represión del AOX en cepas isogénicas con mutaciones en los genes para acetil-CoA sintetasa (acs1, acs2, acs3), isocitrato liasa (icl1) (E.C. 4.1.3.1), malato sintasa (mls1) (E.C. 2.3.3.9), fosfoenolpiruvato carboxiquinasa (pck1) (E.C. 4.1.1.38) y para la enzima málica (mdd1) (E.C. 1.1.1.37), no perjudicaba la represión a causa de la presencia del etanol (66). En contraste, con las mutaciones realizadas en los genes que codifican para las enzimas alcohol deshidrogenasa (adh1, adh2) y acetaldehído deshidrogenasa (aldX), las cuales perjudican la represión inducida por etanol sobre los genes para AOX y catalasa; indicando que el acetato reprime estas dos enzimas. Sibirny sugirió que la molécula de acetato podría ser el co-represor que evoca o promueve la represión de las enzimas encargadas del metabolismo de los compuestos de un carbono (C1) en un medio de cultivo con etanol (37, 66).
Las levaduras P. methanolica y P. pastoris están taxonómicamente muy relacionadas y las vías de utilización de metanol son similares. Esto significa que los genes AOX 1 y AUG1 en P. pastoris y P. methanolica respectivamente, son regulados de manera muy similar. Resultados obtenidos por Inan y Meagher (37), en la expresión de b-galactosidasa (b- gal) en P. pastoris, demostraron que mientras el acetato no fuera consumido no era posible detectar actividad b-gal. Estos autores identificaron dos proteínas que se unen al promotor AOX en extractos celulares de P. pastoris crecidas en las mezclas metanol/etanol y metanol/glicerol y sugieren que alguna de estas proteínas (aún en estudio) podría ser la molécula represora (37).
Algunas de las conclusiones del trabajo de Inan y Meagher (37) sentilde;alaron que concentraciones de acetato y etanol entre 10 y 50mg l-1, inhibían la expresión de b-gal en experimentos realizados en erlenmeyers. agitados, mientras que la adición de 10mg l-1 de acetato y etanol no causaba inhibición en la expresión de b-gal en cultivo alimentado. Lo que demostraba que los niveles de acetato y etanol en bioreactores deben ser monitoreados con detenimiento si se pretende lograr altos rendimientos de la proteína de interés (37).
Poutou et al., (67) estudiando el comportamiento de un clon productor de iduronato-2-sulfato sulfatasa humana recombinante (IDShr) bajo diferentes condiciones de experimentación (67), observaron que prácticamente agotado el glicerol, la adición de 0.5% (v/ v) de metanol generó un aumento en la concentración de O2 disuelto, que en las 40h siguientes se acumulaba el metanol y que la diauxia se extendía hasta las 80h. Fue en este punto donde comenzó el consumo marcado de metanol. En el trabajo de Córdoba (68) se comenta que no se midió la producción de metabolitos como etanol y acetato, sin embargo, teniendo en cuenta el comportamiento en el crecimiento, los resultados son bastante similares a los propuestos por Inan y Meagher (37, 38). En otro de los procesos, para la producción de IDShr, la acumulación de metanol hace que se alcancen niveles de ~5% (v/ v) en el medio de cultivo, lo que ofrece la oportunidad para que la acumulación de H2O2 haya resultado tóxica para la célula, motivo por el cual no se logra recuperar el crecimiento.
En términos metabólicos los resultados obtenidos por Poutou et al., (67) podrían significar que la velocidad de muerte celular (µ(m)) causada por el efecto del H2O2 y no por carencia de sustrato sea igual a la velocidad de crecimiento (µ(x)):
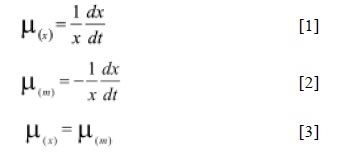
Sin embargo, en este trabajo se manejan los datos de crecimiento como peso seco, lo que no permite discriminar entre las células vivas y las muertas.
Poutou et al., (67) emplearon concentraciones iniciales de glicerol diferentes y en todos los casos la tendencia en la acumulación de metanol fue la misma (40 -100h), lo que hace pensar que alrededor de la hora 80 hay limitación de O2 disuelto o que otros metabolitos como acetato o etanol estaban siendo utilizados hasta entonces para el crecimiento. La duración promedio de los procesos fue de 98.66h y la Actividad específica IDShr presentó un valor promedio de 17.16 nmol/h mg de proteína al final de cada proceso (67, 68), lo que genera incertidumbres como:
¿Hasta que punto, la actividad IDShr detectada al final de los procesos es el resultado de que la velocidad de degradación de la proteínas es igual a la velocidad de síntesis de las nuevas moléculas?
¿Hasta que punto, durante los procesos se ha producido una cantidad considerable de la proteína recombinante, pero una buena parte de ella se produce de manera inactiva y por eso no es detectable por la técnica de actividad enzimática?
La fórmula 3 permite plantear la posibilidad de que las células al morir liberan el contenido intracelular al medio, donde existen proteasas que pueden actuar en su contra. Sin embargo, la concentración de proteínas extracelulares después de las 80h de cultivo es estable (68); y la manera en que justifican este comportamiento es a través de la actividad proteolítica extracelular. En todos los casos, aunque bajas, las mayores actividades proteolíticas aparecieron a las horas finales de los procesos, mostrando cierta relación inversamente proporcional con la actividad de la IDShr.
La segunda incertidumbre se puede resolver siguiendo la producción de la enzima recombinante independientemente de si es o no activa, utilizando anticuerpos policlonales de tipo IgGaIDSh (69) o preferiblemente empleando un ELISA indirecto con anticuerpos IgYaIDSh obtenidos a partir de huevo de gallina, producidos contra péptidos sintéticos que están presentes en epítopes específicos de la IDSh y que no están presentes en ninguna enzima de la misma familia, con lo cual se evita reactividad cruzada entre las enzimas miembro de la misma familia. En este sentido ya existen datos al respecto que serán publicados próximamente (Comunicación personal
Catalina Sosa).
El ácido oléico es un ácido graso de cadena larga mono-insaturado (18:1), e induce la biogénesis de los peroxisomas (70, 71) en varios organismos. Según se mencionó con anterioridad P. pastoris es una de las pocas levaduras capaces de metabolizar metanol y oleato con la consecuente proliferación de los peroxisomas.
Algunos grupos de trabajo han empleado el ácido oléico (CH3 (CH2)7 CH=CH (CH2)7 COOH) como antiespumante gracias a su naturaleza química (67, 68). Sin embargo, son pocos los trabajos donde se haya utilizado este compuesto como única fuente de carbono para el cultivo de P. pastoris, pero se sabe que las levaduras metabolizan el compuesto a través de la b-oxidación, proceso que ocurre únicamente en los peroxisomas (71). La necesidad de procesar el ácido oléico a través de b-oxidación, induce la biogénesis de peroxisomas en las levaduras, lo que genera un aumento en la producción de las enzimas peroxisomales, entre las que se encuentra la AOX.
Al crecer P. pastoris en un medio oleoso, la concen85 tración de AOX es significativa, sin embargo no rebasa el 1 ó 2% de lo que se produce cuando se induce el sistema con metanol (CoMunicación Personal James M. Cregg).
De esta manera, podría deducirse que las enzimas expresadas bajo el control del promotor AOX1 no aumentaran significativamente su concentración por el hecho de utilizar ácido oléico como fuente de carbono. Mientras que la combinación de ácido oleico y glicerol tiene un efecto importante en el aumento de la cantidad de lípidos producidos en las levaduras como S. cerevisiae (72). No obstante, aún no es claro si el mecanismo desencadenado por la presencia de oléico ocurre directamente sobre los promotores de las proteínas peroxisomales (inducción), afecta el contenido de AMPc o desbloquea de alguna manera la represión catabólica que puede ejercer alguna de las otras fuentes de carbono que se estén empleando.
Son varias las estrategias empleadas para el cultivo de P. pastoris con el propósito de expresar proteínas recombinantes. En este artículo se ha discutido el uso de diferentes fuentes de carbono para los cultivos de P. pastoris con el propósito de expresar proteínas recombinantes. Para tal efecto hemos usado como ejemplo nuestra experiencia en la expresión de la enzima IDShr, para la cual hemos logrado niveles importantes de expresión usando inducción con 0.5% (v/v) de metanol cada 24 horas y la conveniencia de seguir la síntesis de la proteína en los cultivos independientemente de la actividad biológica. Hemos discutido la influencia del ácido oleico sobre el sistema de expresión y la necesidad de profundizar en el estudio de los metabolitos secundarios y su acción sobre los niveles de expresión, con miras a optimizar las condiciones de producción de las enzimasrecombinantes en sistemas de expresión AOX1-regulados en P. pastoris.
Agradecemos al Dr. Leonardo Lareo por la revisión crítica del manuscrito y a la Pontificia Universidad Javeriana por el soporte para la realización del estudio (proyectos 1059 y 1714).
1. Cregg J, Barringer K, Hessler A, Madden K. Pichia pastoris as a Host System for Transformations. Mol Cell Biol. 1985; 5 (12): 3376-85.
2. Cregg J, Vedvick T, Raschke W. Recent Advances in the Expression of Foreign Genes in Pichia pastoris. Bio/Technol. 1993; 11: 905-10.
3. Vedvick TS. Gene Expression in Yeast: Pichia pastoris. Curr Opin Biotech. 1991; 2: 742-45.
4. Wolf K. No conventional Yeast in Biotechnology. A Handbook. 1996; Berlin: Springer Verlag. 618 p.
5. Sreekrishna K, Kropp KE. Pichia pastoris, in Nonconventional Yeast in Biotechnology. A Handbook., K Wolf, Editor. 1996, Springer Verlag: Berlin. 203-53 p.
6. Couderc R, Baratti J. Oxidation of Methanol by the Yeast Pichia pastoris: Purification and Properties of Alcohol Oxidase. Agric Biol Chem. 1980; 44: 279-89.
7. Veenhuis M, Dijken JP, Harder W. The Significance of Peroxisomes in The Metabolism of One Carbone Compounds in Yeast. Adv Microbiol Physiol. 1983; 24: 1-82.
8. Córdoba H, Algecira N, Poutou RA, Barrera LA. Pichia pastoris una Alternativa para la Producción de Glicoproteínas Humanas de Uso Terapéutico. Estrategias de Fermentación. Rev Col Biotecnol. 2003; 5 (2): 73-84.
9. Barnett JA. Biochemical Differentiation of Taxa with Special Reference to the Yeast, in The Fungi, GC Ainsworth, AS Sussman, Editors. 1968, Academic Press: London. 557-95p.
10. Barnett JA. The Nutritional Test in Yeast Systematics. J Gen Microbiol. 1977; 99: 183-90.
11. Barnett JA, Payne RW, Yarow D. Yeast: Characteristics and Identification. Cambridge: Cambridge University Press.1990.
12. Kurtzman CP. Synonymy of the Yeast Genera Hansenula and Pichia Demonstrated Through Comparisons of Deoxyribonucleic Acis Relatedness. Antonie van Leeuwenhoek. J Microbiol.1984; 50: 209-17.
13. Spencer JFT, de Spencer ALR, Laluce C. Non-conventional yeasts. App Microbiol Biotechnol. 2002; 58: 147-56.
14. Kato K, Kurimura Y, Makiguchi N, Asai Y. Determination of Strongly Methanol Assimilating Yeast. J Genet App Microbiol. 1974; 20: 123-27.
15. Ogata K, Nishikawa H, Ohsugi M. A. Yeast Capable of Utilizing Methanol. Agricul Biol Chem. 1969; 33: 1319.
16. Invitrogen. Pichia Expression Kit. Protein Expression. A Manual of Methods for Expression of Recombinant Proteins in P. pastoris.Cat. No. K1710-01. 1996; California: Invitrogen.
17. Goul SJ, Mccollum D, Spong AP, Heyman JA, Subramani S. Development of the Yeast Pichia pastoris as a Model Organism for a Genetic and Molecular Analysis of Peroxisomes Assembly. Yeast. 1992; 8: 613-28.
18. Wegner GH. Emerging Applications of the Methylotrophic Yeast. FEMS Microbiol Rev . 1983; 87: 279-84.
19. Raschke W, Neiditch BR, Hendricks M, Cregg JM. Inducible Expression of a Heterologous Protein in Hansenula polymorpha Using the Alcohol Oxidase 1 Promoter of Pichia pastoris. Gene .1996; 177: 163-67.
20. Waterham HR, De Vries Y, Russell YA, Xie W, Veenhuis M, Cregg JM. The Pichia pastoris PER6 Gene Product Is a Peroxisomal Integral Membrane Protein Essential for Peroxisome Biogenesis and Has Sequence Similarity to the Zellweger Syndrome Protein PAF-1. Mol Cell Biol. 1996; 16(5): 2527.36.
21. Gellisen G. Heterologous Protein Production in Methylotrophic Yeasts. App Microbiol Biotechnol. 2000; 54: 741-50.
22. Romanos M. Advances in The Use of Pichia pasrtoris for High-Level Gene Expression. Curr Opin Biotechnol.1995; 6: 527-33.
23. Clare JJ, Rayment FB, Ballnatine SP, Sreekrishna K, Romanos MA. High-level Expression of Tetanus Toxin Fragment C and Pichia pastoris Systems Containing Multiplety Tandem Integrations of the Gene. Bio/Technol.1991; 9: 445-60.
24. Cereghino JL, Cregg JM. Heterologous Protein Expression in the Methylotrophic Yeast Pichia pastoris. FEMS Microbiol Rev. 2000; 24: 45-66.
25. Georgiou G, Valax P. Expression of Correctly Folded Proteins in E. coli. Curr Opin Biotechnol. 1996; 7: 190-97.
26. Li M, Hubálek F, Newton-Vinson P, Edmondson DE. High- Level Expression of Human Liver Monoamine Oxidase A in Pichia pastoris: Comparison with the Enzyme Expressed in Saccharomyces cerevisiae. Prot Exp Purif. 2002; 24: 152- 62.
27. Johnson MA, Waterham HR, Ksheminska GP, Fayura LR, Cereghino JL, Stasyk OV, Veenhuis M, Kulachkovsky AR, Sibirny AA, Cregg JM. Positive Selection of Novel Peroxisome Biogenesis Defective Mutants of the Yeast Pichia pastoris. Genet. 1999; 151: 1379-91.
28. de Hoop MJ, Ab G. Import of Proteins Into Peroxisomes and Other Microbodies. Biochem J. 1992; 286: 657-69.
29. Glover JR, Andrews DW, Subramani S, Rachubinski RA. Mutagenesis of the Amino Targeting Signal of the Saccharomyces cerevisiae 3-ketoacyl-CoA thiolase Reveals Conserved Amino Acids Required for the Import Into Peroxisomes in vivo. J Biol Chem. 1994; 269: 7558-63.
30. Rachubinski RA, Subramani S. How Proteins Penetrate Peroxisomes. Cell. 1995; 83: 525-28.
31. Subramani S. Protein Import Into Peroxisomes and Biogenesis of the Oganelle. Ann Rev Cell Biol. 1993; 9: 445-78.
32. Bohinski RC. Bioquímica. Quinta Edición; Bogotá: Addison- Wesley Iberoamericana.1987.
33. Ren HT, Yuan JQ, Bellgardt KH. Macrokinetic model for methylotrophic Pichia pastoris based on stoichiometric balance. J Biotechnol. 2003; 106: 53-68.
34. Gancedo G, Gancedo JM, Sols A. Glycerol Metabolism in Yeast Pathways of Utilization and Production. Eur J Biochem. 1968; 5: 165-72.
35. Nevoigt E, Stahl U. Osmoregulation and Glycerol Metabolism in the Yeast Saccharomyces cerevisiae. FEMS Microbiol Rev. 1997; 21: 231-41.
36. Ratledge C. Biochemistry and Physiology of Growth and Metabolism, in Basic Biotechnology, C Ratledge, B Kristiansen, Editors. 2001, Cambridge University Press: Cambridge. 17- 45p.
37. Inan M, Meagher MM. The Effect of Ethanol and Acetate on Protein Expression in Pichia pastoris. J Biosc Bioeng. 2001; 92 (4): 337-41.
38. Inan M, Meagher MM. Non-Repressing Carbon Sources for Alcohol Oxidase (AOX1) Promoter of Pichia pastoris. J Biosc Bioeng. 2001; 92 (6): 585-89.
39. Inan M, Chiruvolu V, Eskridge KM, Vlasuk GP, Dickerson K, Brown S, Meagher MM. Optimisation of Temperatureglycerol- pH Conditions for Fed-batch Fermentation Process for Recombinant Hookworm (Ancylostoma caninum) Anticoagulant Peptide (AcAP-5) Production by Pichia pastoris. Enz Microbiol Technol. 1999; 24: 438-45.
40. Chiruvolu V, Cregg JM, Meagher MM. Recombinant Protein Production in an Alcohol Oxidase Defective Strain of Pichia pastoris in Feedbatch Fermentations. Enz Microbiol Technol. 1997; 21: 277-83.
41. Chiruvolu V, Eskridge KM, Cregg JM, Meagher MM. Effects of Glycerol Concentration and pH on Growth of Recombinant Pichia pastoris Yeast. App Biochem Biotechnol. 1999; 75:163-73.
42. Sulter GJ, Klei IJ, Schanstra JP, Harder W, Veenhuis M. Ethanol Metabolism in a Peroxisome Deficient Mutant of the Yeast Hansenula polymorpha. FEMS Microbiol Let. 1991; 82: 297-302.
43. Tolstorukov IDEB, Benevolensky BD, Titorenko VI, Sibirny AA. Mutants of Methylotrophic Yeast Pichia pinus Defective in C2 Metabolism. Yeast. 1989; 3: 233-41.
44. Demain AL, Davies JE. Manual of Industrial Microbiology and Biotechnology; Washintong D.C., USA: Editorial ASM Press. 400. 1996.
45. Murray WD, Duff SJB, Beveridge TJ. Catabolite Inactivation in the Methylotrophic Yeast Pichia pastoris. App Env Microbiol.1990; 56 (8): 2378-83.
46. Egli T, van Dijken JP, Veenhuis M, Harder W, Fiechter A. Methanol Metabolism in Yeast: Regulation of the Syntesis of Catbolic Enzymes. Arch Microbiol. 1980; 124: 115-21.
47. Holzer H. Catabolite Inactivation in Yeast. Trends Biochem Sci. 1976; 1: 176-81.
48. Veenhuis M, Zwart K, Harder W. Degradation of Peroxisomes After Transfer of Methanol Grown Hansenula polymorpha Into Glucose-Containing Media. FEMS Microbiol Let. 1978; 3: 21 28.
49. Veenhuis M, Douma A, Harder W, Osumi M. Degradation and Turnover of Peroxisomes in the Yeast Hansenula polymorpha Induced by Selective Inactivation of Peroxisomal Enzymes. Arch Microbiol. 1983; 134: 193-203.
50. Yuan W, Tuttle D, Shi Y, Ralph G, Dunn W. Glucose-induced Microautophagy in Pichia pastoris Requires the a-subunit of phosphofructokinase. J Cell Sci. 1997; 110: 1935-45.
51. Tuttle D, Dunn W. Divergent modes of autophagy in the methylotrophic yeast Pichia pastoris. J Cell Sci. 1995; 108: 25-35.
52. Yuan W, Strømhaug PE, Dunn WA. Glucose-induced Autophagy of Peroxisomes in Pichia pastoris Requires a Unique E1-like Protein. Mol Biol Cell. 1999; 10: 1353-66.
53. Waterham H, Russell K, De Vries Y, Cregg J. Peroxisomal Targeting, Import, and Assembly of Alcohol Oxidase in Pichia pastoris. The J Cell Biol. 1997; 139 (6): 1419-31.
54. Evers M, Harder W, Veenhuis M. In Vitro Dissociation and Re-assembly of Peroxisomal Alcohol Oxidases of Hansenula polymorpha and Pichia pastoris. FEBS Let. 1995; 368: 293-96.
55. Ellis S, Brust P, Koutz P, Waters A, Harpol M, Gingers T. Isolation of Alcohol Oxidase and two Other Methanol Regulable Genes from the Yeast. Pichia pastoris. Mol Cell Biol. 1985; 5: 1111-21.
56. Cregg J, Madden K, Barringer K, Thill G, Stillman C. Functional Characterization of the Two Alcohol Oxidase Genes from the Yeast Pichia pastoris. Mol Cell Biol. 1989; 9 (3): 1316-23.
57. Koutz P, Davis G, Stillma C, Barringer K, Cregg J, Thill G. Structural Comparison of the Pichia pastoris Alcohol Oxidase Genes. Yeast. 1989; 5: 167-77.
58. Romanos M, Scorer C, Claret J. Foreign Gene Expression in Yeast: A Review. Yeast. 1992; 8: 423-88.
59. Ozimek P, van Dijk R, Latchev K, Gancedo C, Wang D, Van der Klei I, Veenhuis M. Pyruvate Carboxylase Is an Essential Protein in the Assembly of Yeast Peroxisomal Oligomeric Alcohol Oxidase. Mol Biol Cell. 2003; 14: 786-97.
60. Distel B, Veenhuis M, Tabak HF. Import of Alcohol Oxidase Into Peroxisomes of Saccharomyces cerevisiae. EMBO J. 1987; 6: 3111-16.
61. Van der Klei IJ, Veenhuis M, Nicolay K, Harder W. In vivo Inactivation of Peroxisomal Alcohol Oxidase in Hansenula polymorpha by KCN is a Irreversible Process. Arch Microbiol. 1989; 151: 26-33.
62. Van der Klei I, Sulter G, Harder W, Veenhuis M. Assembly of Alcohol Oxidase in the Cytosol of a Peroxisome-Deficient Mutant of Hansenula polymorpha-Properties of the Protein and Architecture of the Crystals. Yeast. 1991; 7: 195-209.
63. Cregg J, Tschopp J, Stillman C, Siegel R, Akong M, Craig W, Buckholz R, Madden K, Kellaris P, Davis G, Smiley B, Cruze J, Torregrossa R, Velicelebi G, Thill GP. High-level Expression and Efficient Assembly of Hepatitis B Surface Antigen in the Methylotrophic Yeast, Pichia pastoris. Bio/Technol. 1987; 5: 479-85.
64. Scorer CA. Rapid Selection Using G1418 of High Copy Number Transformants of P. pastoris for High-Level Foreign Gene Expression. Biotechnol.1994; 12:181-84.
65. Kobayashi K, Kuwae S, Ohya T, Ohda T, Ohyama M, Ohi H, Tomomitsu K, Ohmura T. High-level Expression of Recombinant Human Serum Albumin from the Methylotrophic Yeast Pichia pastoris with Minimal Protease Production and Activation. J Biosci Bioeng. 2000; 89 (1): 55-61.
66. Sibirny AA. Genetic Control of Methanol and Ethanol Metabolism in the Yeast Pichia pinus. in Procedings of 6th International Symposium on Genetics of Industrial Microorganism. Strasbourg: Societe Francese Microbiologie. 1990.
67. Poutou RA, Córdoba H, Quevedo BE, Landázuri P, Echeverri OY, Sáenz H, Vanegas A, Acero J, Gónzalez A, Herrera J, Algesira N, Caicedo L, Barrera LA. Expresión de Iduronato 2-sulfato Sulfatasa Humana Recombinante (IDShr) en Pichia pastoris. Univ Scient. 2005; 10 (1): 73-94.
68. Córdoba H. Estudio de la Producción a Nivel de Laboratorio de la Enzima Iduronato 2-Sulfato Humana recombinante (IDShr) en Pichia pastoris. Facultad de Ingeniería Química. Maestría. Tesis 2004. Universidad Nacional de Colombia. Bogotá, D.C. 97 p.
69. Pentilde;a O, Sosa A, Echeverri O, Sáenz H, Barrera LA. Producción de Anticuerpos Policlonales IgG Contra la Proteína Iduronato-2-sulfato Sulfatasa y Desarrollo de un Sistema de Detección para IDS Humana Recombinante. Bioméd. 2005; 25: 181-88.
70. Cereghino G, Cereghino J, Sunga A, Johnson M, Lim M, Gleeson M, Cregg J. New Selectable Marker/Auxotrophic Host Strain Combinations for Molecular Genetic Manipulation of Pichia pastoris. Gene. 2001; 263: 159-69.
71. Schuller H-J. Transcriptional Control of Nonfermentative Metabolism in the Yeast Saccharomyces cerevisiae. Curr Genet. 2003; 43: 139-60.
72. Dyer JM, Chapital DC, Kuan JW, Mullen RT, Pepperman AB. Metabolic Engineering of Saccharomyces cerevisiae for Production of Novel Lipid Compounds. App Microbiol Biotechnol. 2002; 59: 224-30.