Caracterización molecular de cepas colombianas de Salmonella spp. a través del RFLP-IS200
Elizabeth Vásquez1, Salim Máttar1, Néstor Mossos2, Darío Mogollón2, Raúl A. Poutou3
1Universidad de Córdoba, Instituto de Investigaciones Biológicas del Trópico, Facultad de Medicina Veterinaria & Zootecnia, Montería, Córdoba. Corpoica,2Laboratorio de Diagnostico, 3Laboratorio de Biotecnología Aplicada, Grupo de Biotecnología Ambiental e Industrial, Departamento de Microbiología. Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá, D.C.
Correspondencia: *E-mail: smattar@escarsa.net.co
Recibido:02-05-2005 / Aceptado:26-05-2005
Las técnicas moleculares empleadas para el análisis de ADN cromosomal como el RFLP IS200 han demostrado ser eficientes para resolver relaciones filogenéticas y epidemiológicas entre las cepas de Salmonella spp., aisladas de aves y humanos. El presente estudio tuvo como objetivo determinar si la IS200 estaba presente en las cepas de Salmonella aisladas de aves de corral y de humanos para establecer una posible cadena de transmisión entre ambos. El análisis filogenético de las cepas de Salmonella mostró 13 perfiles de IS200 que exhibieron de 1 a 11 copias de la secuencia de inserción, localizadas en un rango entre 13 y 1.71kb. El trabajo permitió concluir que IS200 es un marcador molecular eficiente, sensible y específico, útil para realizar estudios epidemiológicos ya que discriminó entre cepas de diferentes orígenes y comprobó que existe una relación clonal entre las cepas aviares y humanas. Los resultados obtenidos muestran la necesidad de implementar medidas sanitarias que conlleven a que en la producción se minimicen las condiciones que favorezcan la propagación de Salmonella entre las aves.
Palabras clave: RFLP, Salmonella spp., IS200, dendrograma
Molecular techniques, used for the genomic DNA analysis, as IS200 RFLP has shown to be efficient to solve phylogenetic and epidemiologic relationship between Salmonella spp., strains, isolated from poultry and human. Present study had the aim to determine whether the IS200 sequence was present in the Salmonella spp., strains isolated from both origins, to establish a possible transmition chain between both. Phylogenetic analysis of Salmonella strains showed 13 IS200 profiles that exhibit between 1 and 11 copies of the insertion sequence, located in a range size between 13 and 1.7kb. This work allowed to conclude that IS200 is an efficient, sensible and specific molecular marker, useful to epidemiological studies because it capacity of discriminating between different origin strains and showed the existence of a relationship between this strains.
The results obtained in this work showed the necessity of implementing sanitary rules to minimize the propagation of Salmonella spp., in the poultry production.
Key words: RFLP, Salmonella spp., IS200, dendogram
Salmonella spp. es uno de los miembros más complejos de la familia Enterobacteriaceae con aproximadamente 2400 serotipos descritos hasta la fecha. Actualmente el género se clasifica en dos especies: S. enterica y S. bongori.
En los últimos años se han introducido métodos moleculares de análisis cuyo poder de resolución ha aumentado la posibilidad de diferenciar entre las cepas bacterianas y ha permitido llevar a cabo estudios epidemiológicos más precisos. La comparación de cepas para establecer su identidad se basa en el hecho de que las cepas relacionadas epidemiológicamente provienen de la expansión clonal de un precursor único. Se entiende por clon una cepa que ha sido aislada independientemente de la fuente, en diferentes localizaciones o quizás en diferentes tiempos, pero que muestra caracteres fenotípicos y genotípicos significativamente parecidos, de manera que la explicación más lógica para esta similitud es el origen común (1).
Las técnicas moleculares para el análisis de ADN cromosomal como el RFLP IS200 han demostrado ser un método eficiente para resolver relaciones filogenéticas y epidemiológicas entre cepas de Salmonella spp., aisladas de aves y humanos. En esta técnica la cantidad de copias de la secuencia de inserción (IS) IS200 varía en el genoma de Salmonella spp., entre 1 y 25 copias, la IS200 produce diferentes patrones para los distintos aislamientos de acuerdo a la talla molecular y al número de bandas del elemento genético; de esta manera se puede determinar si dichos microorganismos tienen un origen clonal común (2-5).
El presente estudio tuvo como objetivo determinar si la IS200 estaba presente en las cepas de Salmonella spp., aisladas de aves de corral y de humanos, con el interés de establecer una posible cadena de transmisión entre aves y humanos.
Cepas: entre 1997 y 1998 se recolectaron al azar 260 muestras de materia fecal de pacientes pediátricos con episodios esporádicos EDA en diferentes clínicas de Bogotá. Se recolectaron también muestras de hisopados cloacales, vísceras (hígado, ovario e intestino) y huevos provenientes de explotaciones aviares pertenecientes a zonas de los departamentos de Cundinamarca, Santander, Huila, Boyacá, Meta y Magdalena. Adicionalmente se recolectaron cepas de brotes esporádicos de salmonelosis aviar producidos en Fusagasuga y Bucaramanga en plántas avícolas tecnificadas (Tabla 1).
La identificación bioquímica y serológica de Salmonella spp., se realizó de acuerdo a pruebas de laboratorio estandarizadas previamente. Las pruebas de sensibilidad a los antimicrobianos se realizaron por el método de difusión de Bauer & Kirby (6). Los antimicrobianos evaluados fueron trimetoprimsulfametoxazol (TMP-SXT) [25µg], norfloxacina (nor) [10µg], ampicilina (amp) [10µg], imipenem (imp) [10µg], cloranfenicol (cm ) [30µg] y gentamicina (cn) [10µg] (Oxoid). Extracción de ADN Plásmidico: La extracción de ADN extracromosomal se llevo a cabo a través de la técnica de lisis alcalina descrita por Sambroock et al., (7).
Extracción de ADN cromosomal: Se utilizó la técnica descrita por Ausbel et al., (8).
Análisis de restricción enzimática: Una vez obtenido el ADN cromosomal se llevó a cabo la digestión del mismo con la enzima EcoRI según experiencias previamente descritas (9, 10). La huella genómica producida por la digestión del ADN cromosomal de las cepas de Salmonella spp. fue transferida a una membrana de nylon HybondTM-N+ (Amersham) según el método descrito por Sambroock et al., (7).
Preparación de la sonda: La sonda fue obtenida de la cepa de E.coli DH5a que contenía el plásmido pIZ46 (gentilmente donada por el Dr. Rubino Salvatore de la Universita Degli Studi Di Sassari, Italia).
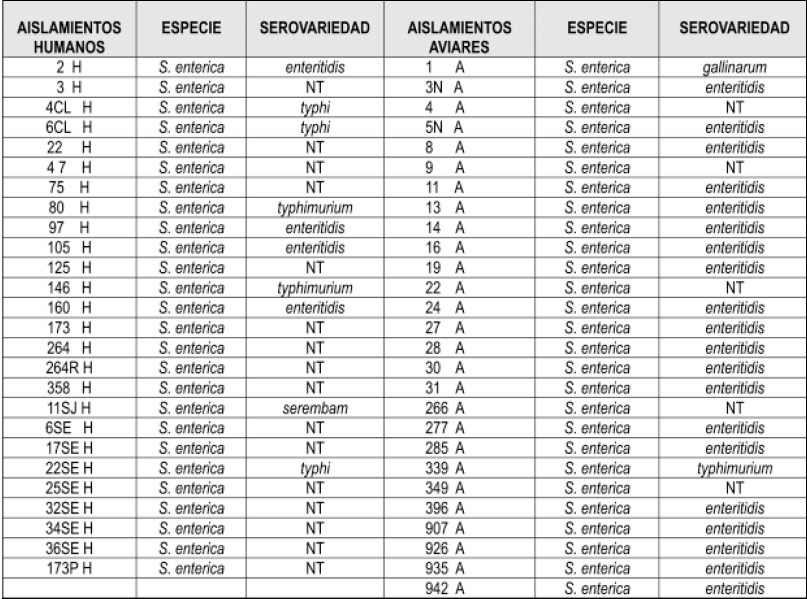
Tabla 1. Cepas de Salmonella spp., de origen humano y aviar NT: cepas no tipificables

Marcaje de la sonda IS200: Para el marcaje de la sonda se utilizó un sistema no radioactivo en el cual se empleó digoxigenina (DIG). La DIG está unida a dUTP por un enlace (debíl) ester-alcalino o un esteroide. El marcaje de la sonda se realizó por PCR con el Kit PCR ELISA DIG-LABELLING (Boehringer Mannheim), para amplificar el monómero de IS200. El volumen final de la mezcla utilizada para la reacción fue 50ul (10X tampón de PCR con MgCl2 DIG, 5ul de 1.25mM dNTPs y 0.35mM DIG-dUTP, pH 7.5 ± 0.2), 4ml de cada iniciador (M13mp18 directo y M13 reverso), 0.5ml de Taq DIG y 1ml del plásmido modificado. Una vez marcada la sonda IS200-DIG se continuó el trabajo para generar los RFLP del ADN cromosomal de las cepas de Salmonella spp., en estudio, utilizando como control positivo la secuencia de inserción IS200 sin marcar y como control negativo la cepa de E.coliATCC 25922.
Interpretación del análisis de restricción: Los patrones fueron considerados diferentes, cuando al menos se detectó una banda polimórfica de ADN. La relación entre las muestras de ADN fue estimada utilizando el coeficiente de Jaccard (11-13).
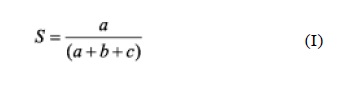
Donde: a es el número de bandas deADN compartidas entre las cepas 1 y 2, b es el número de bandas de ADN presentes en 1 pero no en 2 y c es el número de bandas de ADN presentes en 2 pero no en 1.
Resultados cercanos a 0 indicaron la presencia de patrones de RFLP diferentes (13). Para establecer la distancia genética según el RFLP entre las 55 cepas de Salmonella spp., estudiadas por esta técnica, se construyeron los dendrogramas utilizando una matriz de 0 (ausencia de bandas) y 1 (presencia de bandas) que fue procesada con el programa de computación .Statistica 5.1. de StatSoft, Inc. USA. 1997.
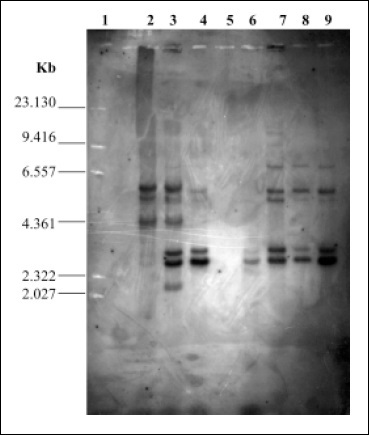
Figura 1. RFLP IS200 de cepas de Salmonella. Distribución de la secuencia de inserción IS200 en algunos de los aislamientos de Salmonella spp. (ADN digerido con EcoRI). Se observa Línea 1: marcador de talla molecular lHind III;Línea 2: cepa SE 22H; Línea 3: cepa 339 A; Línea 4: cepa 396 A; Línea 6: cepa 125H; Línea 7: cepa 285 A; Línea 8: cepa 935 A y Línea 9: cepa 75 H
De las diferentes muestras recolectadas en el campo y en los hospitales se aislaron 55 bacterias con características bioquímicas correspondientes al género de Salmonella spp., 26 cepas se aislaron a partir de muestras humanas y 29 aviares. Después de realizar la identificación serológica de los aislamientos, se determinó que todas las cepas tanto de humanos como de aves correspondieron a Salmonella enterica Tabla 1. El 47.27% de las cepas fueron S. enterica subespecie enterica serovariedad enteritidis, 7.27% S. entérica subespecie enterica serovariedad typhi, 3.63% S. enterica subespecie enterica, serovariedad typhimurium, 1.81% para los serotipos S. enterica subespecie entérica serovariedad gallinarum y S. enterica subespecie enterica serovariedad serembam, el 38.18% restante de las cepas no fueron serotipificables.
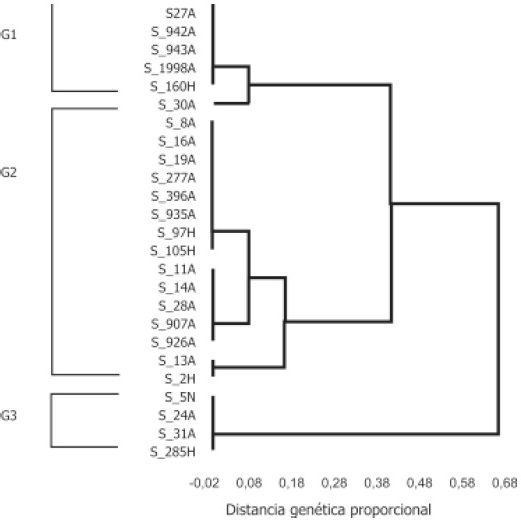
Figura 2. Dendrograma de algunas cepas de Salmonella enterica subespecie enterica serovariedad enteritidis de origen humano y aviar. Grupos G1, G2 y G3.
De otro lado el 61.51% de las cepas de Salmonella aisladas de humanos no fueron serotipificables, el 15.38% se clasificaron como S. enterica subespecie enterica serovariedad enteritidis, 11.54% S. entérica subespecie enterica serovariedad typhi, 7.89% S. enterica subespecie enterica serovariedad typhimurium y el 3.84% restante como S. enterica subespecie enterica serovariedad serembam. Con respecto a las cepas de Salmonella aviares el 75.86% se clasificaron como S. enterica subespecie enterica serovariedad enteritidis, 3.45% S. enterica subespecie entérica serovariedad typhimurium, 3.45% Salmonella entérica subespecie enterica serovariedad gallinarum y el 17.24% de las cepas de Salmonella enterica se clasificaron como no serotipificables Tabla 1 .
El comportamiento de la población de Salmonella spp., demostró que todas las cepas presentaron sensibilidad del 100% frente al imipenem (imp), mientras que el mayor porcentaje de resistencia (7.27%), se presentó contra trimetoprim sulfametoxazol (TMP-SXT) y gentamicina (datos no mostrados).
Es importante resaltar que sólo 3 cepas mostraron resistencia a 2 antibióticos simultáneamente. Además, se observó que la resistencia antimicrobiana presentada por las cepas humanas fue mayor que la evidenciada por las cepas aviares. De las 9 cepas distribuidas entre humanas y aviares que presentaron resistencia a los antibióticos, 4 presentaron un plásmido de talla molecular aproximada a 2.15kb
Las 4 cepas con plásmido resultaron resistentes a TMP-SXT, para verificar la responsabilidad de estos plásmidos frente a la resistencia a TMP-SXT fueron transformados en una cepa de E.coli sensible al antibiótico según describen Poutou et al., (14) (datos no mostrados) y se observó que una vez re-extraídos mostraron la misma talla molecular (2.15kb) y que permitía el crecimiento de la E.coli transformada en agar suplementado con TMP-SXT (50µg/ml), de esta manera se comprobó que la resistencia a dicho antibiótico era mediada por el plásmido aislado, a diferencia de la resistencia a los otros antibióticos de este estudio.
El análisis delADN genómico con la enzima EcoRI mostró que todas las cepas tanto humanas como aviares contenían de 1 a 11 copias de IS200 Figura 1, localizadas en un rango entre 13.013kb y 1.11kb.
Una banda de hibridación de ~2.5kb se presentó en todas las cepas a excepción de la cepa 173 H; sin importar el origen geográfico, el serotipo, ni la procedencia de los aislamientos. Las cepas de Salmonella enteritidis fueron clasificadas en tres grupos (G1, G2 y G3), que al mismo tiempo fueron subdivididos enA, B ó C: el grupo G1- A (cepas 3N, 27 A, 942 A, 943 A, 1998 A y 160H) poseía un perfil característico de dos bandas (2.849 y 2.502kb), el grupo G1-B (cepa 30 A) presentó una banda adicional de 1.147kb. El grupo G2 (15 aislamientos) resultó ser el patrón más representado con 3 subgrupos, G2-A con 5 bandas comunes (2.50, 2.84, 5.27, 6.011 y 7.79kb), el G2-B (cepas 11 A, 14 A, 28 A, 907 A, 926 A y 13 A) conformado únicamente por cepas aviares presentó una banda adicional de 1.86kb, el grupo G2-C (cepa 13 A y 2 H) carecía de la banda de 7.79kb. El grupo G3 estuvo conformado por 4 cepas de origen aviar de distintas regiones de Colombia con un perfil de RFLP IS200 conformado por 4 bandas adicionales con tallas moleculares de 10.10, 11.18, 12.68 y 13.10kb. En la figura 2 se pueden observar algunos de los miembros de los diferentes grupos generados en el análisis.
Este es uno de los primeros trabajos que se lleva a cabo en Colombia sobre epidemiología molecular de Salmonella spp., utilizando como marcador la secuencia de inserción IS200. En el mundo el número de casos de salmonelosis ha aumentado significativamente desde hace dos décadas (15, 16). Aunque diferentes serotipos y fagotipos de Salmonella spp., se han visto involucrados en casos de salmonelosis, el incremento se ha asociado a salmonelosis zoonóticas donde los mayores reservorios han sido los animales de granja (17, 18) por contaminación de los alimentos, pollos y productos cárnicos. Por estos motivos es necesario se ha hecho necesaria la comparación de las cepas de Salmonella spp., aisladas de huevos, carne de pollo y los casos aislamientos de humanos para confirmar si tienen un origen clonal común.
Las técnicas utilizadas para subtipificar y realizar estudios de epidemiología de Salmonella incluyen la serotipificación, el análisis de la resistencia a antibióticos, la fagotipificación (19), el análisis de los patrones de restricción del ADN y el análisis de los perfiles plasmídicos (20-23). En los últimos años la epidemiología molecular se ha convertido en el método más exacto para establecer relaciones filogenéticas entre cepas, debido a que está determinado por genes cromosómicos cuya presencia/ausencia no se ve afectada por condiciones ambientales ni por la presión selectiva de los elementos extracromosomales.
En el presente estudio, entre las Salmonellas tipificables no se encontró ninguna relación entre el origen y el serotipo. S. enterica subespecie entérica serovariedad enteritidis fue el más representado con 47.27% seguido de S. enterica subespecie entérica serovariedad typhi con 7.27%. Estos resultados fueron similares a los obtenidos por González y col. en España (24) con un porcentaje de 52% y 13% respectivamente, pero que difieren de los encontrados en Estados Unidos y en otros países europeos en los que el serotipo más prevalente fue typhimurium (25, 26).
El análisis de las cepas de S. enteritidis con la enzima de restricción EcoRI mostró diferencias interserotipicas evidenciadas por el número de copias de IS200 (de 2 a 9 copias), aunque algunos estudios previos habían reportaron que la secuencia de inserción IS200 no debía ser utilizada para distinguir la relación clonal entre cepas de S. enteritidis, debido al bajo número de copias de (2 a 3 bandas) (27, 28, 4); es importante aclarar que en estos trabajos se utilizó la enzima Pst I para generar la huella genómica del ADN de las cepas de Salmonella.
Este estudio muestra que la enzima EcoRI presenta mayor poder discriminatorio para el análisis de cepas pertenecientes a S. enteritidis. Con respecto a las cepas de S. enteritidis de origen humano (aislamientos 2H, 160H, 97H y 105H) IS200 separó las cepas aisladas en los grupos G1, G2 A y G2B, mostrando que las cepas a pesar de ser fenotípicamente iguales eran diferentes genotípicamente, lo normalmente haría más difícil tanto el tratamiento de la salmonelosis como el control sobre la diseminación de la infección.
Los aislamientos de S. enteritidis de origen humano y aviar fueron caracterizados molecularmente como idénticos por RFLP IS200, sugiriendo que existe una relación filogenética entre los mismos, lo que comprueba la existencia de una cadena de transmisión, por lo que es necesario implementar medidas de control sanitario en las granjas avícolas para prevenir brotes de salmonelosis humana.
Sin embargo, las infecciones aviares no son fáciles de erradicar debido a las condiciones que normalmente se manejan en la mayoría de las granjas; hacinamiento, malas condiciones higiénicas, empleo de incubadoras con aves enfermas, lo que genera la diseminación horizontal (ave-ave) y vertical transovárica (ave-huevo) (29).
En cuanto a las cepas de S. enteritidis aviar se recolectaron cepas de 2 brotes de Salmonella en granjas avícolas de Cundinamarca y Santander; los aislamientos de Santander (277A, 285A, 339A, y 396A) muestran que el brote fue producido por dos serovariedades: S. typhimurium y S. enteritidis, pero dentro de esta última serovariedad se presentaron dos perfiles de RFLP IS200 (Figura 2), lo que sugiere la participación de 2 cepas distintas en dicho brote. Este hallazgo comprobó que la epidemiología molecular es un método eficaz para determinar si un brote es producido
por una sola cepa o no, y de esta manera, implementar medidas de control y prevención de la dispersión del brote.
En el brote de salmonelosis en Cundinamarca se aislaron 6 cepas (907, 926, 935, 942, 943 y 1998) que se identificaron como S. enteritidis pero se clasificaron en dos grupos de RFLP diferentes Figura 2. El resto de las cepas de S. enteritidis de origen aviar pertenecieron a casos esporádicos no relacionados y aislamientos de diferentes regiones del país de salmonelosis aviar pero que son importantes debido a la escasa información que se tiene acerca de estos, aunque constituyen la mayoría de los casos de salmonelosis en nuestro país. Estas cepas se encuentran dentro de los mismos grupos de RFLP IS200 de las cepas de los brotes humanos, sugiriendo que si existen las condiciones para que se favorezca la diseminación y que como consecuencia de ello se podrían presentar nuevos brotes de salmonelosis humana.
Por último es importante resaltar que no se encontró relación epidemiológica alguna entre las zonas avícolas y los patrones de RFLP IS200, lo que demuestra que no existen infecciones causadas por Salmonella localizadas en una determinada región del país, sugiriendo la recirculación de las cepas epidémicas en Colombia quizás debido a la posible utilización de pies de cría infectados. Los 3 grupos de Salmonella enteritidis establecidos en este estudio, no muestran una variedad intraserotípica lo suficientemente grande para inferir que pertenecen a 3 líneas clonales diferentes, sino que las cepas que están circulando tanto en humanos como en aves se encuentran estrechamente relacionados, lo que permitirá en el futuro el desarrollo de técnicas mejores para el control de la salmonelosis en el país.
Es importante resaltar que a pesar de que las cepas aisladas de aves son una fuente de contaminación para los humanos, no son la única vía de diseminación de este microorganismo porque en las cepas de origen humano se aislaron grupos y serovariedades que no estaban presentes en las aves.
En cuanto a la resistencia antimicrobiana a los seis antibióticos, se encontró que la mayor parte (83,64%) eran sensibles a todos ellos, un 10.9% era resistente a un sólo antibiótico y solamente 3 (5.45 %) cepas fueron resistentes a 2 antibióticos. En contraste en un estudio de Hermans et al., en Bangladesh (30), quienes hallaron una resistencia del 96% en las que 67% eran multirresistentes.
En este estudio no puede hablarse de alta resistencia a un antibiótico determinado, ni de una correlación entre serotipo y perfil de resistencia. De todas formas la resistencia antibiótica se consideró de poco valor epidemiológico por el bajo número de cepas resistentes analizadas. Es importante destacar la resistencia de las cepas aviares (10.34%) ya que el uso indebido de antibióticos en medicina veterinaria tiende a generar un incremento en la resistencia antimicrobiana de bacterias patogénicas para humanos, convirtiéndose en un grave problema de salud pública, teniendo en cuenta la naturaleza zoonótica de Salmonella.
Aunque el perfil de plásmidos ha sido previamente utilizado para la epidemiología molecular de Salmonella y de otros microorganismos (31, 14), en este estudio fue imposible establecer alguna correlaci ón epidemiológica usando dicha estrategia debido a que la mayoría de las cepas recolectadas no poseían plásmidos o sólo uno, como ocurrió también en un estudio realizado por Martinelli y Altwegg (32), de esta forma el análisis de las cepas de Salmonella por medio del perfil plasmídico en este estudio mostró un potencial de discriminación limitado por una tasa de aparición plasmídica relativamente baja. En contraste con este trabajo, un estudio de Vatopoulos (33) utilizó el análisis plasmídico para diferenciar 21 cepas de S.enteritidis en 5 grupos distintos.
Aunque los métodos de tipificación como serotipificación, biotipificación, perfiles plasmídicos y resistencia antimicrobiana son utilizados para subtipificar cepas de Salmonella, investigaciones entre cepas de Salmonella aisladas de casos epidemiológicamente relacionados llegan a ser del mismo serotipo. Sin embargo, al realizar pruebas adicionales como sensibilidad a antibióticos y contenido plasmídico son limitados para establecer relaciones epidemiológicas entre subgrupos (31). Por lo tanto, estos marcadores fenotípicos deben ser asociados a marcadores moleculares como el RFLP-IS200, que es una herramienta útil para estudios epidemiológicos y caracterización de aislamientos humanos y aviares, porque proporciona un medio altamente seguro para evaluar la diseminación de las infecciones por Salmonella spp., ya que es posible investigar como una cepa se mueve a través del tiempo entre diferentes regiones o dentro del mismo brote.
Esta investigación muestra que los marcadores fenotípicos y genotípicos deben ser utilizados no únicamente para determinar si los aislamientos son de un mismo clon, si no también para caracterizar cepas para de esta forma identificar reservorios y/o fuentes de contaminación que permiten determinar el origen de la infección. De esta forma se logró demostrar, que IS200 es un marcador molecular eficiente y sensible para estudios epidemiológicos porque discrimina entre cepas de diferentes orígenes y facilita el establecimiento de cierto grado de relación entre cepas aisladas de un origen determinado.
Esta investigación evidenció la necesidad de implementar estudios de epidemiología molecular como el RFLP IS200, en comunidades hospitalarias y poblaciones avícolas para conocer la historia natural de la enfermedad y el seguimiento de las enfermedades infecciosas como también para prevenir futuros casos esporádicos y brotes de salmonelosis que perjudican la salud pública y que tienen graves consecuencias económicas en la industria avícola.
1. Coll P. Interés de las Técnicas de Biología Molecular en el Estudio de las Infecciones Nosocomiales. Microb Clín 1994; 12: 369-371.
2. Beuzón CR, Casadesús J. Conserved Structure of IS200 Elements in Salmonella. Nuc Ac Res 1997; 25: 1355-1361.
3. Casadesús J, Roth J. Absence of Insertions Among Spontaneous Mutants of Salmonella typhimurium. Mol Gen Genet 1989; 216: 210-216.
4. Stanley J, Baquar N, Burnens A. Molecular Subtyping Scheme for Salmonella panama. J Clin Microbiol 1995; 33 (5): 1206- 1211.
5. Stanley J, Saunders N. DNA Insertion Sequences and the Molecular Epidemiology of Salmonella and Mycobacterium. J Med Microbiol 1996; 45: 236-251.
6. Bauer AW, Kirby WMM, Sherris JC, Turck M. Antibiotic Susceptibility Testing by a Standardized Single Disk Method. Am J Clin Pathol 1966; 45: 493-496.
7. Sambrook J, Manniatis T, Fritsch EF. Molecular Cloning: A Laboratory Manual. 2nd ed, ed. T Manniatis, Sambrook, J. Vol. 3. 1989; New York: Cold Spring Harbor Laboratory Press. 459.
8. Ausbel FM, Brent R, Ringston RE, Moore DD, Seidman JG, Smith JA, Struhl R. Current Protocols in Molecular Biology. 1987; New York, USA: Greene Publishing Associates & Wiley Interscience.
9. Olsen JE, Angel O, Threlfall EJ, Bisgaard M. Genomic Relationships Between Selected Phage Types of Salmonella enterica subsp. enterica Serotype typhimurium Defined by Ribotyping, IS200 Typing and PFGE. Microbiol 1997; 143: 1471-1479.
10. Olsen JE, Skov MN, Threlfall EJ, Brown DJ. Clonal Lines of Salmonella enterica serotype enteritidis Documented by IS200-, ribo-, Pulsed-Field Gel Electrophoresis and RFLP Typing. J Med Microbiol 1994; 40: 15-22.
11. Jaccard P. étude Comparative de la Distribution Floral dans une Portion des Alpes et des Jura. Bull Soc Vaudoise Sci Nat 1901; 37: 547-555.
12. Poutou R, Máttar S. Amplificación al Azar del Polimorfismo del ADN (RAPD) de Cepas de E.coli O157:H7 Aisladas de Pacientes Pediátricos. Pediatría 2000; 35 (3): 259-263.
13. Poutou R, Máttar S, Del Portillo P, Visbal J, Bermúdez A. RAPD Fingerprinting of Enterohaemorrhagic Escherichia coli O157:H7 Isolates in Santafé de Bogotá, DC, Colombia. Med Sci Res 2000; 28: 29-32.
14. Poutou RA, Máttar S, Moncayo S, Moreno C. Determinación Molecular De La Multirresistencia En Cepas Aisladas De Infección Nosocomial Del Hospital Universitario San Ignacio De Santafé De Bogotá. MEDICAS UIS 1999; 13 (1): 12- 18.
15. Bögel K. Salmonelosis: An International Comparison. In Symposium .Salmonellose. of German Veterinary Association. 1992. Bonn, Germany.
16. Rodríguez DC, Tauxe RV, Row B. International Increase in Salmonella enteritidis: A New Pandemic? Epid Infect 1990; 105: 21-27.
17. Alterkruse SF, Tollefson LK, Bögel K. Strategies for Salmonella enteritidis in Five Countries. Food Control 1993; 4: 10-16.
18. Talaska T. A Salmonella Data Bank for Routine Surveillance and Research. Bull WHO 1994; 72: 69-72.
19. Oosterom J. Epidemiológical Studies and Proposed Preventive Measures in the Fight Against Human Salmonelosis. Int J Food Microbiol 1991; 12: 41-52.
20. Borrego J, Castro D, Jiménez M, Luque A, Martínez E, Rodríguez A, Picazo J. Comparison of Epidemiological Markers of Salmonella Strains Isolated from Different Sources in Spain. J Clin Microbiol 1992; 30: 3058-3064.
21. Callow BR. A New Phage Typing Scheme for Salmonella typhimurium. J Hyg 1959; 57: 346-349.
22. McDonough Pl, Timoney JF, Jacoson RH, Khakhria R. Clonal Groups of Salmonella typhimurium in New York State. J Clin Microbiol 1989; 27: 622-627.
23. Millemann Y, Lesage MC, Chaslus E, Lafont J. Value of Plasmid Profiling, Ribotyping, and Detection of IS200 for Tracing Avian Isolates of Salmonella typhimurium and S. enteritidis. J Clin Microbiol 1995; 33: 173-179.
24. Gonzáles MA, Alvarez JA, Mendoza MC. Salmonella entérica en el área Sanitaria de Avilés (Asturias). Aspectos Microbiológicos, Clínicos y Epidemiológicos. Enf Infec Microb 1990; 8 (3): 144-147.
25. Cohen ML, Fontaine RE. An Assesment of Patient Related Economic Cost in an Outbreak of Salmonelosis. N Eng J Med 1978; 299: 459-460.
26. Le Minor L, Leminar S, Grimont PAD. Rapport Quatriennal du Center National des Salmonella sur l.origine et la Repartition en Serotypes des Souches Isolees en France Continentale au cours des Annees 1980 a 1983. Rev Epidem Sanite Pub 1985; 33: 13-21.
27. Liebisch B, Schwarz S. Evaluation of Molecular Techniques for Epidemiological Typing of Salmonella enterica subsp. enterica serovar dublin. J Clin Microbiol 1996; 34: 641- 646.
28. Liebisch B, Schwarz S. Moleculat Typing of Salmonella enterica subsp. enterica Serovar enteritidis Isolates. J Med Microbiol 1996; 44: 52-59.
29. Baxter C. Control de la Transmisión Vertical de Salmonella. Avicultura Profesional 1996; 14: 10-12.
30. Hermans PWM, Saha SK, Van Leeuwen WJ, Verbrugh HA, Van Belkum A, Goessens WHF. Molecular Typing of Salmonella typhi Strains from Dhaka (Bangladesh) and Development of ADN Probes Identifying Plasmid-encoded Multidrug-resistant Isolates. J Clin Microbiol 1996; 34 (6): 1373-1379.
31. Brunner F, Margadant A, Peduzzi R, Piffaretti JC. The Plasmid Pattern as Epidemiological Tool for Salmonella typhimurium Epidemics Comparison with the Lysotype. Clin Infec Dis 1983; 148: 711.
32. Martinetti G, Alt W. Egg tRNA Restriction Patterns and Plasmid Analysis as a Tool for Typing Salmonella enteritidis. Res Microbiol 1990; 141: 1151-1162.
33. Vatopoulos AC, Maines E, Balis E, Threlfall J, Kanelopou M, Kalaputhaki V, Malamou H, Legakis MJ. Molecular Epidemiology of Ampicilin Resistant Clinical Isolates of Salmonella enteritidis. J Clin Microbiol 1994; 32: 1322- 1325.