Sandra Mónica Estupiñán Torres MSc.1 y Esperanza Trujillo Gama2
1Docente Programa de Bacteriología, Facultad de Ciencias de la salud, Universidad Colegio Mayor de Cundinamarca. Bogotá, Colombia.
2Fundación Instituto de Inmunología de Colombia
Recibido:23-06-2004; Aceptado:10-09-2004
Sin duda, las vacunas se constituyen en una alternativa viable y efectiva para el control de las enfermedadesinfectocontagiosas, sin embargo, una estrategia exitosa para el diseño de vacunas debe tener en consideración muchos de los aspectos de la respuesta inmune en una población heterogénea y en su forma de reconocer y presentar los antígenos de los patógenos. Por lo tanto, no solamente el entendimiento preciso de los mecanismos inmunes involucrados en la resistencia a infecciones sino la identificación de las moléculas del patógeno que son el blanco de respuesta protectora, serán motivo central de la investigación en vacunas en el futuro inmediato. El presente artículo de revisión profundiza en el descubrimiento, ubicación, expresión, estructura, herencia, polimorfismo e importancia del complejo mayor de histocompatibilidad; además, presenta los avances en elo uso de primates como modelo para el desarrollo de vacunas.
Palabras claves: vacuna, MHC, histocompatibilidad, primates, herencia, desarrollo.
Mayor complex histocompatibility and vaccines development.
Without doubt, the vaccines are the most viable and effective alternative for the control of the contagious infected diseases, however, a successful strategy for the vaccines design must have in consideration many of the aspects of exempt response in a heterogeneous population and in your form of recognizing and presenting the antigens of the patogens. Therefore, not only the accurate understanding of the exempt mechanisms involved in the resistance to infections but the identification of the molecules of the patogen that they are protective response target, they will be central motive of the investigation in vaccines in the immediate future. The present review article deepens in the discovery, location, expression, structure, inheritance, polymorphism and importance of major complex hispocompatibility, and presents the advances in the use of primates as model for the vaccines development.
Key words: vaccine, MHC, histocompatibility, primates, inheritance, development.
Dentro de las funciones más importantes de los linfocitos T están la defensa frente a microorganismos intracelulares y la activación de otras células, como macrófagos y linfocitos B. Una característica esencial para estas funciones es la especificidad limitada de los linfocitos T por los antígenos mostrados por las células del huésped infectadas, las células dendríticas, los macrófagos y los linfocitos B. La tarea de mostrar los antígenos de los microorganismos asociados a células para su reconocimiento por las células T es realizada por proteínas especializadas codificadas por genes situados en un locus denominado Complejo Mayor de Histocompatibilidad (MHC). Así, las moléculas del MHC son parte integral de los ligandos que reconocen la mayoría de las células T, ya que los receptores de antígenos de la célula T son específicos para los complejos de antígenos peptídicos extraños y moléculas del MHC propias.
Bien es sabido que el reconocimiento de antígenos proteicos es primordial para generar una respuesta celular efectora mediada por células T-CD4+, la cual contribuye al control de la infección. Aparte del estudio del MHC del humano, durante las últimas dos décadas, se han adelantado investigaciones en primates no humanos observándose gran homología entre ciertos genes de los dos grupos. En nuestro medio, primates del nuevo mundo de la especie Aotus sp. han sido ampliamente utilizados para la evaluación de antígenos candidatos a vacunas. En nuestro país, se ha utilizado este primate en el proyecto encaminado al diseño de una vacuna contra la malaria causada por el P. falcíparum (1).
1. Descubrimiento: en los años 20, utilizando técnicas clásicas para el estudio de rechazo de tumores y tejidos trasplantados, los investigadores notaron que los injertos de piel de un animal o entre animales de la misma cepa, habitualmente no eran rechazados y los injertos entre animales de diferente cepa eran rechazados con frecuencia, así se estableció la existencia de una base genética para el reconocimiento del injerto como un tejido extraño y a los genes responsables de este fenómeno se les denominó genes de histocompatibilidad.
En los años 60s, se mostró que sueros de mujeres multíparas eran capaces de reconocer moléculas del MHC humano, desde entonces la identificación de los antígenos se ha realizado por técnicas de aglutinación y luego por microcitotoxicidad mediante la activación del complemento. Desde el punto de vista funcional, gracias a los experimentos de Zinkernagel y Doherty (2, 3) en la década de los 70s, se definió la participación crucial del MHC en la presentación de antígenos a los linfocitos T, junto con el reconocimiento de lo propio y lo extraño.
Los fragmentos de longitud variable provenientes de moléculas extrañas o propias al organismo y que ingresan a la célula presentadora del antígeno, son procesados por la maquinaria celular. Con el advenimiento de las técnicas de biología molecular se ha incrementado considerablemente el conocimiento sobre la ubicación de genes y las regiones reguladoras del MHC, así como la tipificación por sondas aleloespecíficas y por secuenciamiento (4 -7). Así, los antígenos del MHC humano inicialmente identificados por técnicas serológicas, han sido luego subdivididos al disponer de sus correspondientes secuencias nucleotídicas.
La constante investigación en el campo de la tipificación de los antígenos del MHC humano, ha permitido el desarrollo de un método denominado RSCA (Reference Strand Conformation Analysis), que ofrece una interesante alternativa para la tipificación de alta resolución. La técnica se basa en la hibridización del exón 2 de la molécula HLA-DRB1 con una sonda específica del exón 2 marcada con fluorescencia, la hibridización es hecha a una temperatura que permite el anillaje aun cuando ocurran malos apareamientos (mismatches). Estos malos apareamientos entre la sonda y el ADN de la muestra forman bucles en el heteroduplex formado. El número y localización de los bucles dará al heteroduplex una movilidad única sobre un gel de poliacrilamida, la asignación de los alelos se hace teniendo en cuenta la movilidad electroforética de los heteroduplex (8).
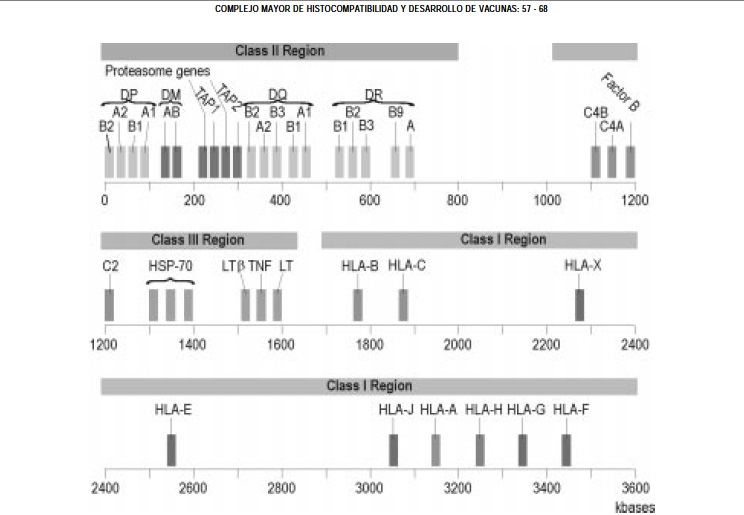
2. Ubicación: el MHC humano se ubica en el brazo corto del cromosoma 6, el complejo abarca cerca de 4 megabases y está dividido en las familias de clase I, II y III, con un número mayor a los 200 genes.

Figura 1. Mapa del MHC humano.
Las moléculas más estudiadas del HLA (Human Leucocyte Antigen) comprenden los antígenos de clase I y II, las cuales tienen rasgos estructurales y funcionales similares, mientras que las moléculas de clase III, cuya función y pertenencia al MHC es aún discutida, son moléculas más heterogéneas.
Las familias de genes clase I, se ubican en loci 11, que contiene 11 familias de genes activos (HLA-DRBA, B, C, E, F y G, MICA, MICB, MICC, MICD y MICE), 4 regiones de pseudogenes (HLA-DRB-H, J, K y L) y otros fragmentos aun no clasificados; los genes de clase II, comprenden el loci 5, DN(O), DM, DP, DQ y DR (9) (Figura 1).
3. Expresión: las moléculas MHC de clase I, se expresan de manera constitutiva en prácticamente todas las células nucleadas, este patrón de expresión esta ligado a las funciones de las células T con restricción por moléculas de clase I del MHC. Debido
a que los virus pueden infectar cualquier célula nucleada, todas las células de este tipo deben presentar los ligandos que reconocen a las células T CD8. Así, proporcionan un sistema de presentación de los antígenos virales.
Las moléculas MHC clase II, presentan péptidos antigénicos generados en la vía endocítica (endosomas y fagosomas) de las APC profesionales (macrófagos, linfocitos B y células dendríticas). Los péptidos presentados por estas moléculas, se fijan al bolsillo de unión con base en interacciones específicas con las cadenas laterales de los aminoácidos que forman el bolsillo de unión del péptido, estos confieren cierta especificidad a cada alelo para fijar determinados péptidos y no otros.
Las moléculas HLA-DR, isotipos de clase II más predominante y polimórfico en el humano (alrededor de 300 haplotipos distintos), juegan un rol fundamental en la selección de linfocitos T CD4+ en el timo y en la periferia en la activación del sistema inmune.
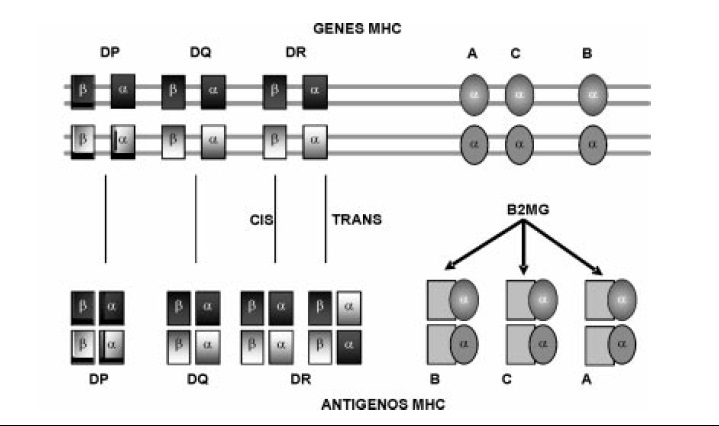
Figura 2. Herencia de las moléculas clase I y clase II del MHC. Para clase I, un individuo heterocigoto expresa seis alelos polimórficos diferentes (tres de cada progenitor) y seis moléculas de clase I por célula. Para la clase II, aunque el individuo también hereda solo seis alelos polimórficos diferentes, cada célula puede expresar más de seis heterodímeros alfa-beta. Se producen heterodímeros adicionales mediante la asociación de la cadena alfa de un alelo y la beta de otro.
La expresión de las moléculas del MHC aumenta por citoquinas producidas durante las respuestas inmunitarias innatas y adaptativas. En casi todos los tipos de células, los interferones alfa, beta y gamma aumentan el grado de expresión de las moléculas de clase I; el factor de necrosis tumoral y la linfotoxina también pueden tener este efecto.
La expresión de las moléculas de clase II, también esta regulada por citoquinas diferentes en células distintas. El interferón gamma es la principal citoquina implicada en la estimulación de la expresión de las moléculas de clase II, especialmente en monocitos y macrófagos.
4. Herencia: los genes del MHC se expresan de manera codominante en cada individuo, es decir, cada individuo expresa los alelos del MHC en los dos cromosomas heredados de los padres. Para el individuo, esto aumenta al máximo el número de moléculas del MHC disponibles para ligar péptidos y presentarlos a las células T (10).
En estudios previos de cartografiado genético, se ha encontrado que puede haber más de dos genes funcionales de cadena beta para algunos loci clase II, pero habitualmente solo uno para cadena alfa. El uso de más de un gen de cadena beta permite que algunos genes de clase II especialmente DR sean expresados en más de dos formas alélicas por una sola célula, esto contribuye a marcar una gran diferencia entre los genes de clase I y clase II; para clase I un individuo heterocigoto expresa seis alelos polimórficos diferentes (tres de cada progenitor) y seis moléculas de clase I por célula. Para la clase II, aunque el individuo también hereda solo seis alelos polimórficos diferentes, cada célula puede expresar más de seis heterodímeros alfa-beta. Se producen heterodímeros adicionales mediante la asociación de la cadena alfa de un alelo y la beta de otro (Figura 2) (10).
El conjunto de alelos del MHC presentes en cada cromosoma se denomina haplotipo del MHC. En los seres humanos, cada alelo da HLA recibe una designación numérica. Por ejemplo, un haplotipo de HLA de un individuo puede ser HLA-A3, HLA-B5, HLA-DR52, entre otros. Todos los individuos heterocigotos tienen, por supuesto, dos haplotipos de HLA. También en seres humanos, algunos alelos de HLA que se encuentran en distintos loci se heredan juntos con mayor frecuencia de la que cabría esperar por azar, fenómeno denominado desequilibrio de ligamiento.
5. Polimorfismo: las moléculas clase I y clase II del Complejo Mayor de Histocompatibilidad, presentan un alto polimorfismo con varios cientos de variantes alelicas de algunos de los genes en la población. El polimorfismo es encontrado predominantemente en los dominios alfa 1 y 2 y beta 1, de las moléculas de clase I y de clase II respectivamente (Figura 3). Estos dominios proteicos forman el sitio de unión a péptido en cada caso, permitiendo a estas moléculas unir diversos péptidos.
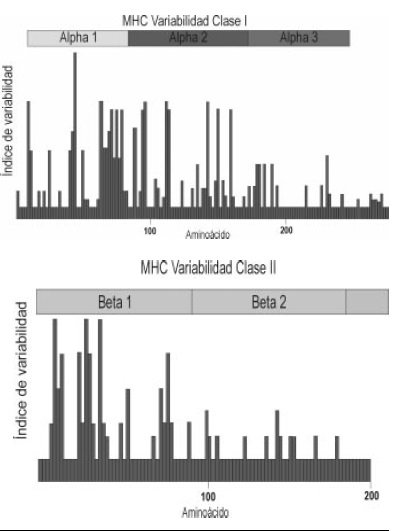
Figura 3. Polimorfismo del MHC. A. Polimorfismo MHC clase I. La mayor variabilidad en aminoácidos en las diferentes posiciones a lo largo de la cadena alfa de las moléculas del MHC clase I ocurre en las regiones alfa 1 y alfa 2. El mayor polimorfismo se encuentra en los aminoácidos que forman la pared y el piso del sitio de unión a péptido. B. Polimorfismo MHC clase II. El mayor polimorfismo para la cadena beta de las moléculas clase II se encuentra en los
aminoacidos de la región beta 1, que forma la pared y el piso del sitio de unión al antígeno.
Una de las características más sobresalientes de las moléculas de clase II, es su amplio polimorfismo; las diferencias más destacadas del polimorfismo del HLA-DRB con el polimorfismo de otros loci nucleares, están el gran número de alelos por locus, la gran variabilidad entre alelos y la mayor diversidad en los loci que codifican para las cadenas beta o pesadas en comparación a los que codifican las cadenas alfa o livianas.
Dicho polimorfismo es inusual, ya que se ha mantenido por una selección balanceada y no por una selección neutra, la cual es mucho más común en otras familias multigénicas dentro de poblaciones naturales. Mediante esta selección balanceada se explica la frecuencia intermedia de una gran cantidad
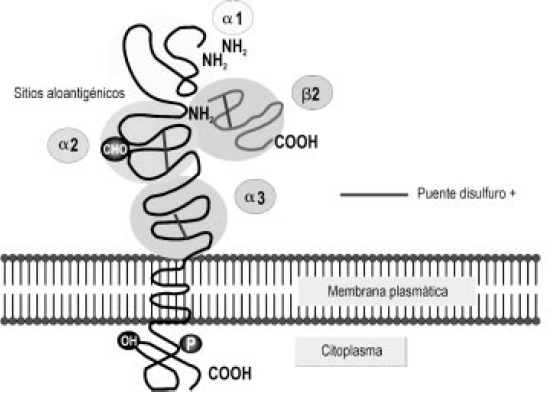
Figura 4. Estructura de la molécula de MHC clase I. La molécula de MHC clase I tiene tres dominios globulares alfa 1, alfa 2 y alfa 3. El dominio alfa 3 está estrechamente relacionado con la beta 2 microglobulina.
de alelos, favorecida entre otras razones, por una selección sobredominante, en la cual la tasa de sustituciones no sinónimas (con reemplazo de aminoácidos) en la PBR, sobrepasa la de sinónimas (sin reemplazo de aminoácidos).
Por otra parte las regiones no codificantes como los segmentos intergénicos, los intrones y las regiones 3 no traducidas (3UTR), aparentemente estarían siendo seleccionadas por mutaciones neutras, pero su polimorfismo podría deberse en parte a su cercanía con las regiones codificantes (11). Múltiples evidencias apoyan la hipótesis según la cual la principal fuerza seleccionante en el MHC y que permite su diversidad alélica, es la ventaja conferida a las moléculas del complejo para unir una gran variedad de péptidos, su presentación restringida y específica al sistema inmunológico, y la probable resistencia a múltiples patógenos.
6. Estructura: el esclarecimiento de las características bioquímicas de las moléculas del MHC ha sido uno de los logros más importantes de la inmunológica moderna. El avance clave en este campo fue la resolución de las estructuras cristalinas de las porciones extracelulares de las moléculas de clase I y clase II (12,13), posteriormente se han cristalizado y analizado en detalle numerosas moléculas de MHC unidas a peptidos. Basándonos en este conocimiento actualmente entendemos la base
del funcionamiento de las moléculas del MHC para la presentación de péptidos.
En las moléculas de clase I, cada familia génica codifica para dos cadenas peptídicas heterodiméricas denominadas cadena pesada (cadena alfa) y liviana (Beta 2 microglobulina, codificada fuera del complejo), la cadena pesada (44 a 47 kD) se orienta de manera que alrededor de las tres cuartas partes del polipéptido completo se extienden en el medio extracelular, un pequeño segmento atraviesa la membrana y el extremo carboxi terminal se localiza en el citoplasma (Figura 4).
Los segmentos alfa 1 y alfa 2 del extremo amino terminal interaccionan y forman el sitio de unión al péptido, su tamaño es suficientemente grande para unir peptidos de 8 y 11 residuos de aminoácidos en una conformación flexible y extendida, los extremos de esta hendidura están cerrados de manera que peptidos de mayor tamaño no pueden encajar (14) (Figura 5).
Las moléculas de clase II, están compuestas por dos cadenas polipeptídicas unidas de forma no covalente una cadena alfa (no polimórfica) y cadena beta (polimórfica). A diferencia de lo que sucede en las moléculas de clase I, las dos cadenas de la molécula de clase II están codificadas por genes polimorficos del MHC. Los extremos alfa 1 y beta 1 del extremo amino terminal de las moléculas de clase II interaccionan y forman la hendidura de unión al péptido, estructuralmente similar a la de clase I (Figura 6), en las moléculas de clase II, los extremos de la hendidura de unión al péptido están abiertos, de manera que pueden unirse peptidos de 10 a 20 residuos (Figura 7) (14).
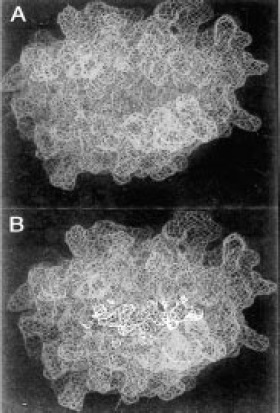
Figura 5. Sitio de unión al antígeno de la molécula de clase I. A. Los segmentos alfa 1 y alfa 2 del extremo amino terminal interaccionan y forman el sitio de unión al péptido. B. Sitio de unión a péptido con péptido unido, su tamaño es suficientemente grande para unir péptidos de 8 a 11 residuos de aminoácidos en una conformación flexible y extendida, los extremos de esta hendidura están cerrados de manera que péptidos de mayor tamaño no pueden encajar.
Los estudios cristalográficos de varias moléculas clase I y II, revelan que la asociación péptido-MHC se presenta por la interacción débil entre las cadenas laterales de los péptidos y una región de las moléculas formada por las cadenas alfa y beta del heterodímero la cual se denomina hendidura de unión al péptido.
Esa región es polimórfica y presenta unos sitios “bolsillos” en los cuales encajan perfectamente las cadenas laterales del péptido unido. Ese polimorfismo es el causante de que un mismo péptido se una con diferente afinidad a las diferentes clases de moléculas MHC. Los bolsillos se ubican en regiones bien definidas de la hendidura y se han identificado para las moléculas de clase II con números como P1, P4, P6 y P9, mientras que estos son denominados con letras para las moléculas de clase I (14) (Figura 8).
Así mismo, los residuos de anclaje (aminoácidos dentro del péptido que son esenciales para el acoplamiento del péptido a las moléculas de MHC) son variables para cada molécula; estos residuos de anclaje determinan si la unión a las moléculas del MHC es o no favorable para que el complejo antígeno – MHC sea reconocido por el sistema inmune y genere una respuesta inmune efectiva (Figura 9).
La capacidad de las moléculas MHC clase II para unir péptidos antigénicos fue demostrada inicialmente por trabajos como los de Buu, aunque un alelo de MHC puede interactuar con una amplia gama de determinantes antigénicos, la unión es selectiva y no todos se unen con igual afinidad (15). Estos estudios también revelan que las cinéticas de formación y disociación del complejo son inusuales con una tendencia a ser lentas requiriendo horas para ser detectadas.
Investigaciones hechas sobre la formación del complejo péptido-MHC clase II han identificado un intermediario presente en los minutos iniciales de la reacción que precede a la formación del complejo final, más estable (16).
7. Importancia: la importancia médica clínica del MHC se centra fundamentalmente en el campo de los transplantes y en las asociaciones de ciertos HLA con enfermedades. En los transplantes de órganos como riñón, hígado, corazón, pulmón, cornea, entre otros, la tipificación del MHC se usa rutinariamente. De manera sobresaliente se ha documentado que los transplantes de médula ósea, corazón y pulmón, requieren un análisis pormenorizado del HLA-DRB, así como de los antígenos menores de histocompatibilidad. Estas tipificaciones detalladas intentan
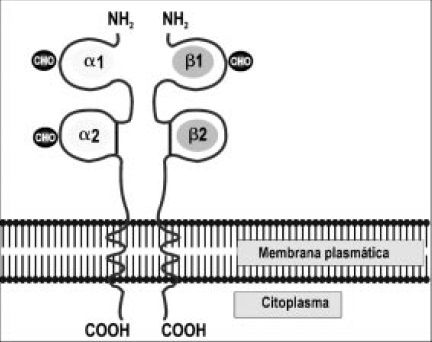
Figura 6. Estructura de la molécula MHC clase II. La molécula de MHC clse II está compuesta por dos cadenas polipeptídicas diferentes (alfa y beta) las cuales están asociadas de manera no covalente
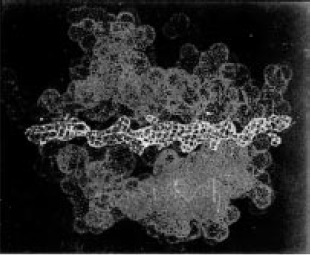
Figura 7. Sitio de unión al antígeno de la molécula de clase II. Los extremos alfa 1 y beta 1 del extremo amino terminal de las moléculas de clase II interaccionan y forman la hendidura de unión al péptido, estructuralmente similar a la de clase I. En las moléculas de clase II, los extremos de la hendidura de unión al péptido están abiertos, de manera que pueden unirse péptidos de 10 a 20 residuos.
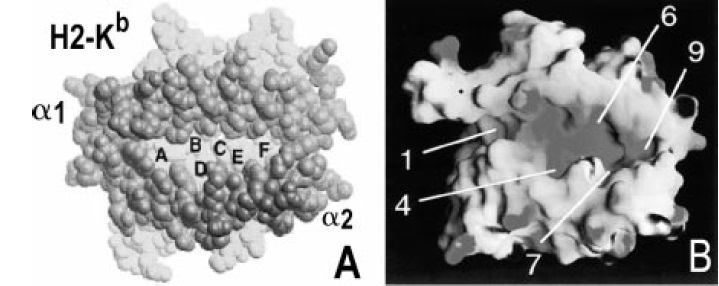
Figura 8. Bolsillos de unión de las moléculas clase I y clase II del MHC A. Bolsillos de unión de la molécula de clase I. En estas moléculas los bolsillos son numerados con letras de la A a la F. B. Bolsillos de unión de las moléculas clase II. En estas moléculas los bolsillos son numerados: 1, 4, 6, 9.
reducir los posibles rechazos, mejorando la sobrevida post-transplante (17, 19).
Algunas enfermedades humanas ocurren con mayor frecuencia en individuos portadores de determinados alelos de los genes del MHC. Entre estas se encuentran las enfermedades autoinmunes, la susceptibilidad a agentes infecciosos (20) y algunos otros procesos clínicos (21,22). Sin embargo, en ninguna de estas condiciones la asociación estadística es absoluta, lo cual indica que algunos otros genes, así como factores ambientales, desempeñan su papel en el mecanismo fisiopatogénico de las mismas. Es así como la mayor asociación HLA-enfermedad conocida, la presenta la espondilitis anquilosante con el HLA B27. El 90 % de los pacientes caucásicos con esta enfermedad expresan el mencionado alelo, frente al l 9 % de la población control (22).
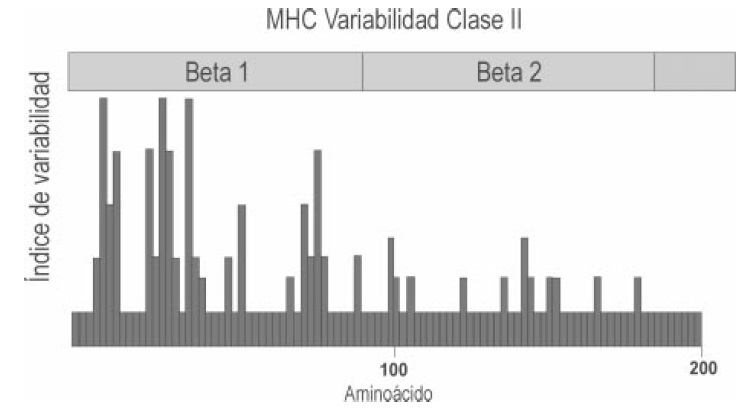
Figura 9. Unión de péptidos a las moléculas del MHC. A. Comparación de la unión de péptidos a una molécula clase I, HLA-A2, y a una molécula de clase II, HLA-DR1. B. Localización de los bolsillos del MHC y los residuos de anclaje de los péptidos en diferentes complejos péptido- molécula de clase II del MHC
8. MHC y diseño de vacunas sintéticas: la identificación de epitopes que conducen a la efectiva activación de Linfocitos T ayudadores y citotóxicos, es el fin principal de los nuevos candidatos a vacunas, dada la simplicidad para su síntesis, su bajo costo y su capacidad
– solas o en combinación con vacunas de ADN
– para generar respuesta inmune contra microorganismos complejos.
Las vacunas basadas en péptidos químicamente fabricados (vacunas sintéticas), serán una importante alternativa para la inmunoprofilaxis de las enfermedades infecciosas en un futuro. Una epitope ayudadora o Th ideal deber ser “universal”, es decir que sea capaz de interactuar con un gran número, sino con todos los halotipos de MHC clase II. Aunque la interacción de péptidos antigénicos con moléculas extremadamente polimórficas como las de clase II representa uno de los mayores obstáculos para la identificación de epítopes universales y diseño de vacunas sintéticas, muchos trabajos recientes por ejemplo: (i) estudios de cristalografía de rayos X han definido las características del bolsillo de unión, mostrando preferencias entre alelos para su interacción con cadenas laterales de los aminoácidos en los péptidos; (ii) la generación de datos experimentales, de ensayos de unión in vitro entre péptidos y moléculas MHC purificadas (23); (iii) la caracterización a través del mapeo de proteínas y péptidos de respuestas de células efectoras especificas contra péptidos; (iv) la identificación de residuos críticos para la interacción del péptido con el receptor de las células T para el antígeno (TCR) y (v) el reciente aporte de la informática al campo, que ha hecho posible la identificación de “motivos universales” y matrices virtuales HLA clase II, lo anterior ha permitido mejor comprensión de las interacciones entre péptidos y estas moléculas, y quizás conlleve en un futuro, a esclarecer adecuadamente las características de posibles epitopes universales.
9. Primates no humanos como modelo para el desarrollo de vacunas: el estudio del MHC humano, en las últimas dos décadas, y las investigaciones en primates no humanos, han permitido observar gran homología entre ciertos genes de los dos grupos. Las equivalencias alélicas se han documentado a lo largo de las especies, lo anterior de acuerdo con la hipótesis de trans-especie, según la cual, los genes actuales del MHC han evolucionado a partir de alelos ancestrales comunes a varias especies.
Cada familia génica dentro de una especie particular se ha especializado, aunque manteniendo en ocasiones rasgos estructurales y funcionales (24), los genes varían tanto intra como interespecie presentándose diferentes grados de polimorfismo alélico y del haplotipo entre cada una de ellas. De manera interesante se ha observado que la velocidad de aparición de nuevos genes, se mantiene relativamente constante en la escala evolutiva. Por esta razón, se postula que el MHC ha sufrido varios procesos de duplicación, seguidos por contracciones génicas en el transcurso de millones de años. Esta modalidad adaptativa le ha permitido al complejo ajustarse, manteniendo regiones y polimorfismos antiguos con capacidad de responder a microorganismos anteriores o actuales y generando regiones nuevas frente a nuevas especies de microorganismos (25).
Paralelo a la exploración de la biología de primates no humanos y otros mamíferos con fines académicos o de preservación de la biodiversidad, ha surgido la necesidad apremiante de emplear diversas especies de monos para estudiar enfermedades humanas. De resaltar ha sido el uso de chimpancés y de monos Rhesus en estudios de protección frente al virus de inmunodeficiencia humana (HIV), así como el de monos Aotus en enfermedades como hepatitis B, malaria, esquistosomiasis y últimamente en enfermedades autoinmunes como la artritis reumatoide y esclerosis múltiple (26,27).
Diversos estudios, desde el punto de vista biológico y biomédico, han permitido describir analogías del MHC de primates no humanos con el HLA. De tal manera, se conoce que existen homologías génicas del MHC (ortologías) entre los monos del viejo mundo (Catarrhini) y el humano. Sin embargo, las analogías con los primates del nuevo mundo (Platyrrhini) y el humano o de los Platyrrhini con los Catarrhini son menos claras. Las diferencias interespecie en el complejo radican en la funcionalidad de ciertos alelos, la aparición de variantes nuevas y la inactivación de genes (pseudogenes) (28,29).
Aunque se conoce con mayor detalle lo que ha pasado con el MHC de otras especies de monos del nuevo mundo, no hay hasta la fecha estudios poblacionales sobre el MHC de Aotus, y solo dos grupos científicos han descrito algunos alelos de clase I y II en muy pocos animales (30-34).
Los monos Aotus son unos de los primates más antiguos en el nuevo mundo y su distribución geográfica es amplia; por ello es deseable descifrar en detalle cual o cuales serían los posibles vínculos evolutivos del Aotus con otras especies y con el humano. De igual forma no hay hasta la fecha reportes que describan cuales son las reglas de procesamiento antigénico o de presentación inmunológica empleadas por el sistema inmune de este animal, especialmente si se tiene en cuenta que el Aotus es una especie ampliamente empleada como modelo de enfermedades humanas.
El origen del MHC se remonta a millones de años antes de la aparición del Homo sapiens. Por ello, su estudio y comprensión, permiten realizar inferencias evolutivas sobre el origen de grupos de animales, dadas sus características de polimorfismo trans-especie, cuyos elementos funcionales han sido adaptados por muy diversas especies.
En nuestro medio, primates del Nuevo mundo de la especie Aotus spp han sido ampliamente utilizados par la evaluación de antígenos candidatos a vacunas (1). En nuestro país, se ha utilizado este primate en el proyecto encaminado al diseño de una vacuna contra la malaria causada por el P. falcíparum (1). Los promisorios resultados obtenidos en estos trabajos, hacen necesario esclarecer los mecanismos de respuesta inmune frente a las vacunas, y aquellos que median la protección.
Para ello, durante los últimos años y mediante estrategias de biología molecular, se ha logrado establecer la secuencia del receptor de las células T a-b y g-d (35), de las Interleuquinas 2, 4, 6,10 (36), la secuencia de la cadena liviana kappa genes C, V y J (37-42) y de algunos genes del Complejo Mayor de Histocompatibilidad (homólogos de las moléculas clase I y del HLA-DQ, DP y DR del humano) (30-33), las investigaciones muestran una estrecha relación estructural con su contraparte en humano.
Desde los puntos de vista estructural y funcional, la caracterización de péptidos exógenos con capacidad de unión a varios alelos (promiscuidad de unión) que se hallan presentes en distintas poblaciones (43- 47) y su posible asociación con la resistencia o susceptibilidad a enfermedades infecciosas, son una herramienta muy poderosa para diseñar vacunas sintéticas en las que se combinan fragmentos de longitud variable (48-51). Mediante esta estrategia se busca asegurar una protección duradera y efectiva frente a microorganismos patógenos en cualquier población.
1. Patarroyo ME, Romero P, Torres ML, Clavijo P, Moreno A, Martínez A, et al. Induction of protective immunity against experimental infection with malaria using synthetic peptides. Nature 1995; 328: 629-32.
2. Zinkernagel RM, Doherty PC. Immunological surveillance against altered self-components by sensitized T lymphocytes in lymphocytic choriomeningitis. Nature1974; 215: 547-8.
3. Doherty PC, Zinkernagel R. Enhanced immune surveillance in mice heterozygous at the H-2 gene complex. Nature 1975; 256: 50-2.
4. Bugawan TL, Begovich AB, Erlich HA. Rapid HLA-DPB typing using enzymatically amplified DNA and nonradioactive sequence-specific oligonucleotide probes. Immunogenetics 1990; 32: 231-41.
5. Vilches C, García-Pacheco JM, De Pablo R, Puente S, Kreiser M. Complete coding region of the new HLA-DQB1*0612 allele, obtained by RT-PCR. Tissue Antigens 1996; 48: 589-92.
6. Craig MA, Elliot JF. HLA-DQA1 genotyping by bi-directional sequencing of PCR-amplified DNA spanning exon 2. Tissue Antigens 1997;49: 417-20.
7. Buyse I, Decorte R, Baens M, Cuppens H, Semana G, Emonds MP, et al. Rapid DNA typing of class II HLA antigens using the polymerase chain reaction and reverse dot blot hibridization. Tissue Antigens 1993; 41: 1-14.
8. Lobashevsky A, Smith J, Kasten-Jolly J, Horton H, Knapp L, Bontrop RE, et al. Identification of DRB alleles in rhesus monkeys using polymerase chain reaction-sequence-specificprimers (PCR-SSP) amplification. Tissue Antigens 1999; 54:254-63.
9. McIlhatton B, Keating C, Curran M, McMullins M, Barr J, Madrigal A, et al. Identification of medically important pathogenic fungi by reference strand-mediated conformational analysis (RSCA). J. Med. Microbiol 2002; 51:468-78.
10. Abbas A, Lichtman A. Complejo principal de histocompatibilidad. En: Inmunologia cellular y molecular. España: McGraw Hill Interamericana; 2000. p: 66-81.
11. Villadangos JA. Presentation of antigens by MHC class II molecules: getting the most out of them. Molecular Immunology 2001; 38: 329-46.
12. Mach B, Steimle V, Martinez-Soria E, Reith W. Regulation of MHC class II genes: Lessons from a disease. Annu.Rev.Immunol 1996; 14: 301-31.
13. Bowers, W. Major Histocompatibility Complex (MHC), Disponible en: URL: http://www.med.sc.edu:85/bowers/mhc.htm
14. Andersson G, Andersson L, Larhammar D, Rask L, Sigurdardóttir S. Simplifying genetic locus assignment of HLA-DRB genes. Immunology Today 1994; 15: 58-62
15. Gyllensten UB, Erlich HA. MHC class II haplotypes and linkage, disequilibrium in primates. Human Immunolgy 1993; 36: 1-10.
16. Anderssson G. Evolution of the human HLA-DR region. Frontiers in Bioscience 1998; 3: 739-45.
17. Klein J. Origin of major histocompatibility complex polymorphism: The trans-species hypothesis. Human Immunology 1987; 19: 155-62.
18. Bjorkman PJ, Saper MA, Samraoui B, Bennett WS, Strominger JL, Wiley DC. Structure of the human class I histocompatibility antigen HLA-A2. Nature 1987; 329: 2506-12.
19. Stern JL, Brown JH, Jardetzky TS, Gorga JC, Urban RG, Strominger JL, et al. Crystal structure of the human class II protein HLA-DR1 complexed with an influenza virus peptide. Nature 1994; 368: 215-21.
20. Dessen A, Lawrence M, Cupo S, Zaller D, Wiley DC. X-ray structure of HLA-DR4 (DRA*0101, DRB1*0401) complexed with a peptide from human collagen II. Immunity 1997; 7: 473-81.
21. Smith K, Pyrdol J, Gauthier L, Wiley DC, Wucherpfennig KW. Crystal structure of HLA-DR2 (DRA*0101, DRB1*1501) complexed with a peptide from human myelin basic protein. J. Exp. Med 1998; 188: 1511-20.
22. Paul WE. Fundamental immunology. Chapter 8. Lippincott: Williams & Wilkins; 1999.
23. Engelhard VH. Structure of peptides associated with class I and class II molecules. Annu. Rev. Immunol 1994; 12: 181-207.
24. Buus S, Sette A, Colon S, Miles C, Grey H. The relation between major (MHC) restriction and the capacity of Ia to bind immunogenetic peptides. Science 1987; 235: 1353 - 7.
25. Sadegh-Nasseri S, Stern L, Wiley D, Germain R. MHC class II function preserved by low-affinity peptide interactions preceding stable binding. Nature 1994; 370: 647-50.
26. Calmus Y. Graft-versus-host disease following living donor liver transplantation: high risk when the donor is HLA-homozygous. J Hepatol 2004; 41:505-7.
27. Laux G, Mytilineos J, Opelz G. Critical evaluation of the amino acid triplet-epitope matching concept in cadaver kidney transplantation. Transplantation 2004; 77:902-7. COMPLEJO MAYOR DE HISTOCOMPATIBILIDAD Y DESARROLLO DE VACUNAS: 59 - 70
28. Schaffer M, Aldener-Cannava A, Remberger M, Ringden O, Olerup O. Roles of HLA-B, HLA-C and HLA-DPA1 incompatibilities in the outcome of unrelated stem-cell transplantation. Tissue Antigens 2003; 62: 243-50.
29. Hill AV, Bennett S, Allsopp CE, Kwiatkowski D, Anstey NM, Twumasi P, et al. HLA, malaria and dominant protective associations. Parasitol Today 1992; 8:57.
30. Liu C, Carrington M, Kaslow RA, Gao X, Rinaldo CR, Jacobson
LP, et al. Lack of Associations Between HLA Class II Alleles and Resistance to HIV-1 Infection Among White, Non-Hispanic Homosexual Men. J Acquir Immune Defic Syndr 2004; 37:1313- 17.
31. Fanning LJ, Kenny-Walsh E, Shanahan F. Persistence of hepatitis C virus in a white population: associations with human leukocyte antigen class 1. Hum Immunol 2004; 65:745-51.
32. Balamurugan A, Sharma SK, Mehra NK. Human leukocyte antigen class I supertypes influence susceptibility and severity of tuberculosis. J Infect Dis 2004; 189:805-11.
33. Inmunogenética en las enfermedades reumáticas. En: Reumatología pediátrica. Interamericana: McGraw-Hill; 1994. p 21-36.
34. Zavazava N, Eggert F. MHC and behavior. Immunol Today 1997; 18 (1):8-10.
35. Vargas LE, Parra CA, Salazar LM, Guzman F, Pinto M, Patarroyo ME. MHC allele-specific binding of a malaria peptide makes it become promiscuous on fitting a glycine residue into pocket 6. Biochem Biophys Res Commun 2003; 307 (1):148-56.
36. Klein J, Sato A, O’huigin C. Evolution by gene duplication in the major histocompatibility complex. Cytogenet. Cell. Genet 1998; 80: 123-7.
37. Voss G, Manson K, Montefiori D, Watkins DI, Heeney J. Prevention of disease induced by a partially heterologous AIDS virus in rhesus monkeys by using an adjuvanted multicomponent protein vaccine. J Virol 2003; 77:1049-58.
38. Gramzinski RA, Brazolot Millan CL, Obaldia N, Hoffman SL, Davis HL. Immune response to a hepatitis B DNA vaccine in Aotus monkeys: A comparison of vaccine formulation, route, and method of administration. Mol. Med1998; 4: 109-18.
39. Colllins WE. The owl monkey as a model for malaria. In Aotus: the owl monkey. Baer JF, Weller RE and Kahoma I. editors. New York: Academic Press; 1994. p. 217-244.
40. Antunes SG, De Groot NG, Brok H, Doxiadis G, Menezes AL. Otting N, et al. The common marmoset: a New World primate species with limited MHC class II variability. Proc. Natl. Acad. Sci 1998; 95: 11745-50.
41. Trtkova K, Kupfermann H, Grahovac B, Mayer WE, O’huigin C, Tichy H, et al. MHC-DRB genes of Platyrrhini primates. Immunogenetics 1993; 38: 210-22.
42. Suarez CF, Cardenas PP, Llanos-Ballestas EJ, Martinez P, Obregon M, Patarroyo ME, et al. Alpha1 and alpha2 domains of Aotus MHC class I and Catarrhini MHC class Ia share similar characteristics. Tissue Antigens 2003; 61:362-73.
43. Díaz-Arévalo D, Naegeli M, Rodriguez R, Niño-Vasquez JJ, Moreno A; Patarroyo ME, et al. Sequence and diversity of MHC-DQA and –DQB genes of Aotus nancymaae, a primate model for vaccine development. Immunogenetics 2000; 51:528-532
44. Diaz D, Daubenberger CA, Zalac T, Rodriguez R, Patarroyo ME. Sequence and expression of MHC-DPB1 molecules of the New World monkey Aotus nancymaae, a primate model for Plasmodium falciparum. Immunogenetics 2002; 54:251-9.
45. Niño-Vásquez JJ, Vogel D, Rodríguez R, Moreno A, Patarroyo ME, Pluschke G,et al. Sequence and diversity of DRB genes of Aotus nancymaae, a primate model for human malaria. Immunogenetics 2000; 51: 219- 30.
46. Vecino W, Daubenberger C, Rodrigue ZR, Moreno A, Patarroyo M, Plushcke G. Sequence and diversity of T-cell receptor bchain V and J genes of the owl monkey Aotus nancymaae. Immunogenetics 1999; 49: 792-99.
47. Guerrero JE, Pacheco DP, Suarez CF, Martinez P, Aristizabal F, Moncada CA, et al. Characterizing T-cell receptor gammavariable gene in Aotus nancymaae owl monkey peripheral blood. Tissue Antigens 2003; 62:472-82.
48. Hernández EC, Suárez CF, Méndez JA, Echeverri SJ, Murillo LA, Patarroyo ME. Identification, cloning, and sequencing of different cytokine genes in four species of owl monkey. Immunogenetics 2002; 54: 645-53.
49. Diaz O, Daubenberger CA, Rodríguez R, Naegeli M, Moreno A, Patarroyo ME, et al. Immunoglobulin kappa light-chain V, J, and C gene sequences of the owl monkey Aotus nancymaae. Immunogenetics 2000; 51: 212-18.
50. Castillo F, Guerrero C, Trujillo E, Delgado G, Martinez P, Salazar LM, et al. Identifying and structurally characterizing CD1b in Aotus nancymaae owl monkeys. Immunogenetics 2004; 56: 480-89.
51. Montoya GE, Vernot JP, Patarroyo ME. Comparative analysis of CD45 proteins in primate context: owl monkeys vs. humans. Tissue Antigens 2004; 64:165-72