Frecuencias alélicas de 14 STR’s autosómicos en una población de Antioquia, Colombia
Humberto Ossa Reyes1, Edna Patricia Tascón Peñaranda1, Hugo Andrés Moreno Chaparro1, Jorge Horta Salinas2, Gustavo Moreno Rodríguez1
1Laboratorio de Genética y Biología Molecular, Bogotá, Colombia.
2Universidad El Bosque. Facultad de Biología, Bogotá, Colombia
Correspondencia: hossa@cable.net.co
Recibido: 03-05-2010 / Aceptado: 06-01-201
Resumen
En esta investigación se analizaron 50 individuos independientes de una muestra total de 5´671.689 habitantes correspondientes al Departamento de Antioquia en las ciudades de Medellín, Envigado, Sabaneta y Santa Fe de Antioquia, con el fin de realizar la huella genética. Los STR autosómicos utilizados fueron CSF1PO, TH01, TPOX, D16S539, D7S820, D13S317, F13A01, vWA, HPRTB, D8S1179, D5S818, PENTA E, D18S51 y D3S1358. Con los datos obtenidos de las frecuencias alélicas se analizó el equilibrio de Hardy–Weinberg, el índice de fijación y algunos parámetros forenses, mediante los programas Genepop Versión 3.2 y PowerState. Se observó que la población no presentó diferencias significativas para la prueba de Hardy – Weinberg y en su confirmación con la prueba de F. Los resultados médicos forenses mostraron un índice de discriminación acumulado de 0,999992947, el índice de exclusión estuvo por encima del 0,9906 y la probabilidad de coincidencia acumulada fue de 1 en 7,05311E-06 individuos.
Palabras clave: equilibrio Hardy – Weinberg, frecuencias poblacionales, huella genética, STR’s autosómicos.
Abstract
Allele frequencies of 14 STR’s autosomal in a population of Antioquia, Colombia
En esta investigación se analizaron 50 individuos independientes de una muestra total de 5´671.689 habitantes correspondientes al Departamento de Antioquia en las ciudades de Medellín, Envigado, Sabaneta y Santa Fe de Antioquia, con el fin de realizar la huella genética. Los STR autosómicos utilizados fueron CSF1PO, TH01, TPOX, D16S539, D7S820, D13S317, F13A01, vWA, HPRTB, D8S1179, D5S818, PENTA E, D18S51 y D3S1358. Con los datos obtenidos de las frecuencias alélicas se analizó el equilibrio de Hardy–Weinberg, el índice de fijación y algunos parámetros forenses, mediante los programas Genepop Versión 3.2 y PowerState. Se observó que la población no presentó diferencias significativas para la prueba de Hardy – Weinberg y en su confirmación con la prueba de F. Los resultados médicos forenses mostraron un índice de discriminación acumulado de 0,999992947, el índice de exclusión estuvo por encima del 0,9906 y la probabilidad de coincidencia acumulada fue de 1 en 7,05311E-06 individuos.
Palabras clave: equilibrio Hardy – Weinberg, frecuencias poblacionales, huella genética, STR’s autosómicos.
Introducción
Los STR’s, repeticiones cortas en tándem, que se encuentran distribuidos a lo largo del genoma de los organismos eucariotas, aunque en una baja frecuencia en regiones codificantes y en los telómeros (1), han representado gran utilidad en el estudio de la huella genética humana, en el esclarecimiento de su reciente historia evolutiva y para pruebas de filiación biológica (2).
Los 14 STR’s autosómicos utilizados poseen un elevado poder de discriminación, es decir, una
heterocigocidad superior al 90%, una ubicación cromosómica diferente, de tal manera que se evite la selección del loci cercano que pueda conjugarse como un grupo de ligamiento, un bajo índice de mutación y un rango de longitud alélica de 90-500 pb (3).
Ya que Colombia posee un alto grado de diversidad génica, con este trabajo se pretende aportar al campo investigativo una base de datos con frecuencias alélicas que se podrá aplicar para estudios poblacionales, de tipo forense e identificación humana con base en una muestra de 50 individuos independientes de una región de Antioquia.
Materiales y métodos
Se seleccionó una muestra de la población antioqueña en los municipios de Envigado, Sabaneta
y Santa fe de Antioquia localizados al noreste de Colombia. Se escogieron 50 individuos independientes teniendo en cuenta los 50 apellidos de mayor frecuencia en la población Antioqueña (4, 5), a los cuales se les confirmó su origen, lugar de nacimiento, ascendencia por parte materna y paterna y un consentimiento informado, el cual firmó cada individuo que participó en el estudio. La extracción de ADN se realizó mediante la técnica de Salting-Out (6), varias PCR múltiplex (7) y por último la tipificación alélica de cada individuo.
Posteriormente se obtuvieron las frecuencias alélicas, algunos estadísticos de variabilidad genética, el equilibrio de Hardy – Weinberg mediante el programa Genepop versión 3.4 (8, 9,
10) y los parámetros estadísticos de interés legal calculados por PowerState (1, 11, 12).
Resultados
Al analizar los datos obtenidos se presentó una distribución de 27 mujeres (54%) y 23 hombres
(46%). A cada muestra se le asignó su respectivo alelo proveniente del linaje materno y paterno. Con base en las frecuencias alélicas obtenidas, Tabla 1 y Figura 1, se puede observar que la población se comportó de manera normal, ya que no presentó diferencias significativas. El sistema Penta E fue el marcador que presentó un mayor número de alelos entre los individuos y D3S1358 fue el marcador que presento un menor número de alelos.
Para los marcadores vWA y Penta E solo se tipificaron 48 muestras, y para D18S51 49, debido a las bajas concentraciones de ADN extraído para estas muestras. En el caso del marcador HPRTB se observó que los alelos 12 y 13 presentaron una frecuencia muy alta, ya que son homocigotos. Con referencia a los marcadores HPRTB y D16S539 se encontraron diferencias significativas para el equilibrio de Hardy-Weinberg, y después de la correlación fueron excluidos, ya que los alelos encontrados fueron extremadamente homocigotos, lo cual incidió en el análisis.
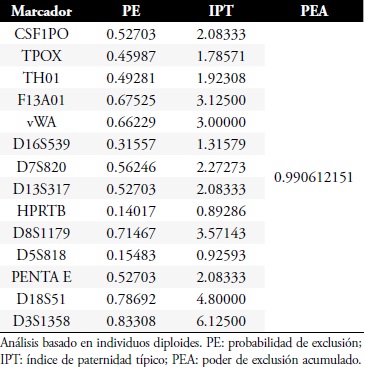
Con base en las frecuencias observadas y en los resultados tanto de los heterocigotos como homocigotos esperados para el cálculo de Hardy- Weinberg se obtuvo un promedio de diversidad genética de 0.766580±0.389410, demostrando una población con comportamiento normal. También se calcularon los datos de ámbito forense, encontrándose un poder de discriminación acumulado de 0.999992947 y un poder de coincidencia acumulado de 1 en 7.05311E-06, Tabla 2. Además se calcularon los datos de poder de exclusión acumulado arrojando un valor de 0.990612151, Tabla 3.
Discusión
Se puede observar que la población dentro de cada rango de alelos se comportó de manera normal, ya que no se encontraron diferencias significativas entre ellos. Al presentar un mayor número de alelos para el marcador Penta E se demuestra que la población antioqueña se encuentra en un alto grado de mestizaje, esto debido a las migraciones que ha tenido la región en el pasado, haciendo posible la presencia de nuevos alelos o el flujo genético (13).
Con respecto a la tipificación de HPRTB se observó la presencia de homocigotos, dado que es un marcador del cromosoma X (14), y como se obtuvo un 46 % de hombres en la población estudiada, estos solo presentarían un solo alelo, mientras que la otra parte de la población tipificó dos alelos para cada mujer muestreada. Como se eliminó el HPRTB y el D16S539 por las diferencias significativas para el equilibrio de Hardy-Weinberg, se analizaron finalmente 12 marcadores por el método de chi-cuadrado, y se aceptó la hipótesis nula del equilibrio de Hardy-Weinberg, describiendo a esta población con reproducción sexual aleatoria, sin selección y mutaciones.
Tabla 1. Frecuencias alélicas de 14 STR’s de una región de Antioquia
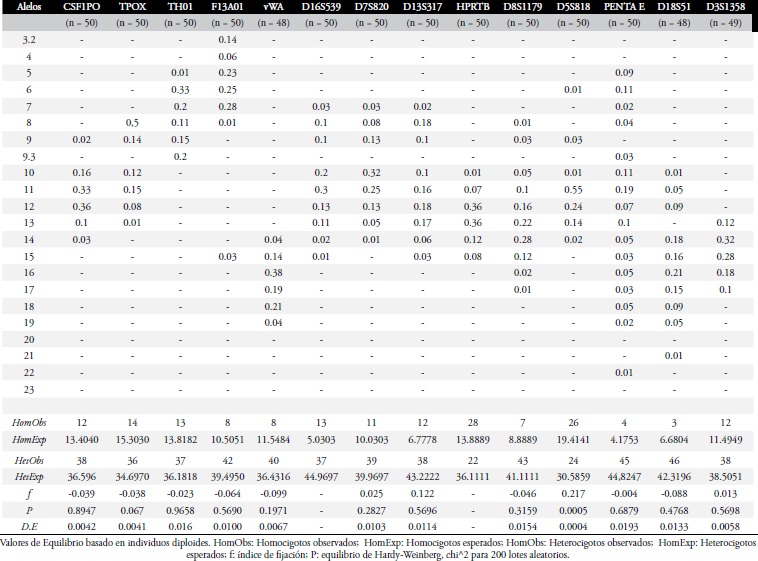
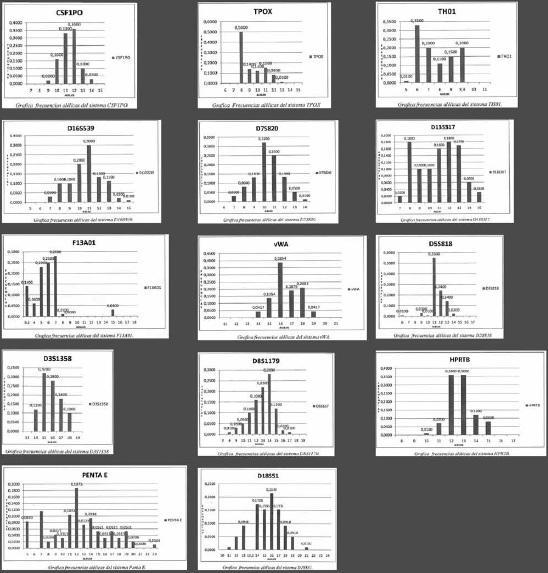
Figura 1. Frecuencias alélicas de los 14 STR´s
El promedio de diversidad genética obtenido demuestra que la población se encuentra dentro del comportamiento normal de los heterocigotos y homocigotos esperados (9). Teniendo en cuenta los datos forenses encontrados, como el poder de discriminación acumulado y el poder de coincidencia acumulado, estos valores ofrecen un alto grado de confiabilidad para la identificación humana y forense (11,12). Con base en los resultados obtenidos se puede observar que la población, a pesar de encontrase en aislamiento geográfico, no se encuentra en un aislamiento genético, ya que mediante los sistemas de ADNmt y cromosoma Y estudiados por Bedoya en 2006 (15), se ha desarrollado un flujo genético, el cual ha caracterizado los 14 STR`s estudiados.
Tabla 2. Valores Forenses
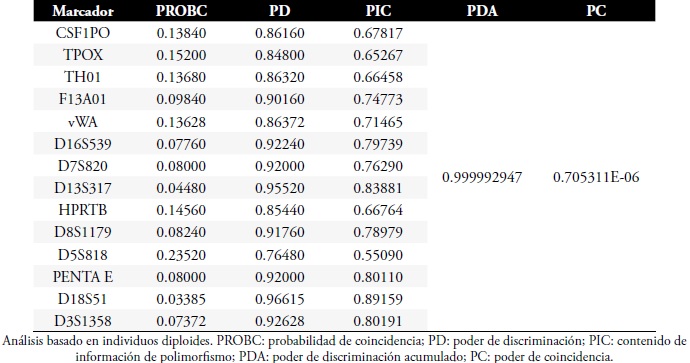
Tabla 3. Análisis de paternidad
Agradecimientos
A la población antioqueña y al Laboratorio de Genética y Biología Molecular por su aporte para poder realizar esta investigación.
Referencias
1. Butler J, M. Biology, Technology and Genetics of STR Markers. Forensic AND typing 2005. Second edition; 273.
2. Méndez A. J, Bravo R, Isea W and Jordana J. Microsatellites (STR´s), AND Molecular Markers for Excellency. Arch Latinoam Prod Anim 2005; 30-42.
3. Gill P, Sparkes R and Kimpton C. Development of Guides to designate alleles using an STR multiplex. Forensic Science International 1997; 82: 185-197.
4. álzate N. La Bella Villa. Medellín, Colombia, la Casa del Cristiano. Primera edición. 2000. 5. Argos R. Historia de Antioquia. Medellín, Colombia. Dirección de Cultura de Antioquia. 1996.
6. Miller S. A, Dykes D. D and Polesky H. F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Research 1988; 16 (3): 1215.
7. Lodish H, Berk A, Zipursky L, Matsudaria P y Baltimore D. Biología Celular y Molecular. Cuarta edición. Mexico: Editorial Medica Panamericana 2003.
8. Cavalli-Sforza L. L. y Bodmer W. F. Genética de las poblaciones humanas 1981. Ediciones Omega. 956.
9. Gou S. L and Thompson E. Performing the Exact test of Hardy – Weinberg propotion for multiple alleles. Biometric 1992; 48: 361-372.
10. Weir B and Cockerham C. Estimating f-statistic for the analysis of population structure. Evolution 1984; 1358-1370.
11. González J. F. Análisis Molecular de Variación de Polimorfismos STR autosómicos y de cromosoma Y en grupos étnicos de Ecuador con aplicación médico-forense [tesis doctoral]. Zaragoza: Departamento de Anatomía Patológica, Medicina Legal, Forense y Toxicología, Universidad de Zaragoza; 2006.
12. Goodwin W, Lanacre A and Hadi S. An Introduction to Forensic Genetics. England. Jhon Wilwy & Sons Ltda. 2007. 75-85.
13. Bravo L and Builes J. Analysis of 12 STR loci in Antioquia (Colombia popular sample. International Congress Series, 151-153.
14. GenBank [Base de Datos Internet]. National Center for Biotechnology Information. 1982- [fecha de acceso 23 de Abril 2010]. Disponible en: http://www.ncbi.nlm.nih.gov/
15. Bedoya G, Montoya, P, García J, Soto I, Bourgeois S and Carvajal L. Admisture Dynamics in Hispanics: A Shift in the Nuclear Genetic Ancestry of a South American Population isolate. Proc Natl Acad Sci USA (79): 7234-7239.




